Homology analysis of Acinetobacter baumannii in intensive care unit before and after COVID-19 pandemic
-
摘要:
目的 探究新型冠状病毒感染大流行前后上海市部分医疗机构重症监护病房(ICU)环境中病原菌的分布及鲍曼不动杆菌同源性变化情况。 方法 采用基质辅助激光解析电离飞行时间质谱(MALDI-TOF MS)法分离鉴定病原菌,通过Illumina Miseq测序平台对鲍曼不动杆菌进行全基因组测序,基于多位点序列分型(MLST)和核心基因组MLST(cgMLST)探讨其亲缘关系。 结果 大流行后ICU环境中病原菌总检出率(6.10%,101/1 656)低于大流行前(10.77%,181/1 680;P<0.05)。大流行前后,ICU环境中检出的鲍曼不动杆菌在被褥表面占比均维持在最高水平,但大流行后ST型呈多样性分布。MLST_Pasteur结果显示,162株鲍曼不动杆菌分为20种ST型,以ST2(80.25%,130株)为主;MLST_Oxford结果显示,162株菌株共有19种ST型,以ST208(37.04%,60株)为主;基于cgMLST的聚类分析结果显示,大流行后ST208_Oxford与ST540_Oxford和ST369_Oxford的亲缘关系更接近,ST164_Pasteur克隆由大流行前的ST234_Oxford转变为大流行后的ST1418_Oxford,新发现2种新型ST_Pasteur和11种新型ST_Oxford。 结论 大流行后ICU环境中病原菌检出率低于大流行前,大流行前后相同分离位点ST型分布略有不同,大流行前后的流行克隆仍以ST2_Pasteur/ST208_Oxford为主,但部分等位基因发生了改变,cgMLST在同源性分析、进化与传播、暴发分析方面的精确度优于MLST_Oxford和MLST_Pasteur。 Abstract:Objective To investigate the distribution of pathogens and change of homology of Acinetobacter baumannii (AB) in intensive care units (ICUs) of partial medical institutions in Shanghai before and after the pandemic of coronavirus disease 2019 (COVID-19). Methods Pathogens were isolated and identified by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF MS), the whole genome of AB was sequenced using Illumina Miseq sequencing platform, and its genetic relationship was explored based on multilocus sequence typing (MLST) and core genome multilocus sequence typing (cgMLST). Results The total detection rate of pathogens in ICU environment after the pandemic was lower than before the pandemic ([6.10%, 101/1 656] vs [10.77%, 181/1 680]; P < 0.05). Before and after the pandemic, the proportion of AB detected on the surface of bedding in the ICU environment remained at the highest level. After the pandemic, the ST types showed a diverse distribution. MLST_Pasteur results showed that 162 strains of AB were divided into 20 ST types, with ST2 (80.25%, n=130) being the main type. MLST_Oxford results showed that there were 19 ST types among 162 strains, with ST208 (37.04%, n=60) being the main type. The clustering analysis based on cgMLST showed that ST208_Oxford had closer genetic relationship with ST540_Oxford and ST369_Oxford after the pandemic. ST164_Pasteur clone changed from ST234_Oxford before the pandemic to ST1418_Oxford after the pandemic, and 2 new types of ST_Pasteurr and 11 new types of ST_Oxford were discovered. Conclusion The detection rate of pathogens from ICU environment after the pandemic is lower than before the pandemic, and the distribution of ST types is slightly different at the same detection sites before and after the pandemic. ST2_Pasteur/ST208_Oxford is still the dominant epidemic clone before and after the pandemic, but some alleles have changed. cgMLST is more accurate than MLST_Oxford and MLST_Pasteur in homology analysis, evolution, spread, and outbreak analysis. -
Keywords:
- COVID-19 /
- Acinetobacter baumannii /
- ST type /
- homology analysis /
- intensive care unit environment
-
新型冠状病毒感染(COVID-19,简称新冠感染)大流行是1918—1920年西班牙流感以来世界卫生组织承认的最大的大流行[1]。新冠感染大流行期间,医院重症监护病房(ICU)是抢救、治疗的主要单元,但也是引起医院感染的主要源头[2],而鲍曼不动杆菌(Acinetobacter baumannii, AB)是ICU中最常见,易造成患者感染,甚至引起医院感染暴发的病原菌[3],西班牙、墨西哥和巴西等医院均报道过医院ICU患者COVID-19和AB的双重感染[4-6];以色列一所医院也报道[7]过在COVID-19患者中的AB感染暴发事件。医院ICU正逐渐成为AB滋生的主要场所,其干燥表面已被证实是医院大流行传播的重要源头。全球公认巴斯德(Pasteur)和牛津(Oxford)两种针对AB的多位点序列分型(MLST)方案,前者包括cpn60、fusA、gltA、pyrG、recA、rplB和rpoB基因,后者依赖于gltA、gyrB、gdhB、recA、cpn60、gpi和rpoD基因。两种方案有3个共同的管家基因,在进化分析方面各有利弊,目前多以巴斯德方案为主[8]。我国17省的研究[9]显示,AB具有较高流行率,ST2型是我国及全球最主要的流行克隆。近期研究[10]发现,ICU中ST2型AB逐渐向ST164型克隆演化,并在ICU患者和环境中广泛传播。另一方面,ICU是医院感染防控的重点场所,尤其在新冠感染大流行后,医院的运营与管理方式发生了显著变化,精细化感染防控制度[11]、三级网络管理体系[12]等干预措施使医院感染防控、消毒等工作得到了全面加强和提升。为了解新冠感染大流行防控策略对ICU环境病原菌检出及其同源性分布的具体影响,本研究统计分析了上海市部分医疗机构新冠感染大流行前后常见病原菌的检出情况,并以AB为研究对象探讨其同源性变化,以期对后续医疗机构的感染防控、消毒举措等提供数据支持。
1. 材料与方法
1.1 标本来源
根据上海市及浦东新区耐药菌监测方案,按照GB 15982—2012《医院消毒卫生标准》采样方法分别采集2019、2023年医疗机构综合ICU的环境物体表面和工作人员手标本。用浸有采样液的无菌棉签在物体表面来回涂抹5次,剪掉手接触部位后将采样棉签放入装有10 mL采样液的采样管中,其中物体表面<100 cm2时取涂抹全部表面,≥100 cm2时涂抹100 cm2,手涂抹面积约为30 cm2/只。环境物体包括诊疗器械(呼吸机等)、诊疗相关用品(治疗推车等)、工作人员直接接触用品(工作服等)、患者直接接触物品(被褥等)、保洁用品(抹布等)、共用用品(水龙头等)6类标本。在各医疗机构未对ICU环境进行彻底消毒前进行标本采集,采样频率为每所医疗机构每月每类标本采集2~3份,2023年未采集到约束带、空调进出风口和病号服标本,其余采样点和2019年保持一致。
1.2 分组情况
2019年共纳入7所医疗机构(编号:A~G)的ICU为标本采集点,每所医疗机构采集80~440份标本,共检测1 680份标本,纳入大流行前组;2023年除上述7所医疗机构外,还新增医疗机构(编号:H)的ICU为标本采集点,每所医疗机构采集180~356份标本,共检测1 656份标本,纳入大流行后组。
1.3 仪器与试剂
飞行时间质谱仪(德国布鲁克公司),测序仪(美国Illumina公司)、大豆酪蛋白消化卵磷脂吐温肉汤(Soya Casein Digest Lecithin Polysorbate Broth, SCDLP)、胰酪大豆胨液体培养基(Tryptic Soy Broth, TSB)等增菌液(英国OXOID公司),AB显色平板、哥伦比亚血平板等(上海科玛嘉公司),DNA提取试剂盒(西安天隆科技公司),建库试剂盒(美国Illumina公司)等。
1.4 方法
1.4.1 菌株分离与鉴定
标本接种到SCDLP(用于金黄色葡萄球菌和铜绿假单胞菌的增菌、培养)和TSB(用于AB和肺炎克雷伯菌的增菌、培养)培养基中,(36±1)℃培养48 h后,分别划线接种于相应的显色平板和血平板上,(36±1)℃培养24~48 h,挑取可疑菌落进行革兰染色、氧化酶等生化试验,并通过基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)鉴定进行二次确证,评分值>2.1即为目标菌株,将纯菌落保存在3%甘油脑心浸液肉汤中,-80℃保存。
1.4.2 DNA提取
将保存于-80℃冰箱中的AB划线接种于血平板上,37℃培养24 h后,挑取圆形、灰白色、光滑的纯菌落进行质谱鉴定,评分值>2.1即为目标菌株。用无菌接种环刮取3~5个纯菌落转移到盛有500 μL生理盐水的EP管中并制成菌悬液,按照西安天隆科技有限公司的磁珠法核酸提取试剂盒说明书提取基因组DNA后,使用Qubit DNA定量试剂盒检测DNA浓度,浓度≥10 ng/μL即可进行全基因组测序。
1.4.3 全基因组测序
按照Illumina DNA prep试剂盒说明书依次进行DNA片段化、扩增、纯化、均一化等步骤,将文库稀释至4 nmol/L起始浓度,最终上机浓度为12 pmol/L。通过Miseq测序仪进行测序,应用CLC数据分析工具对下机数据进行质控、组装,并在BLAST网站(Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast.cg)对组装结果进行菌种确证。
1.4.4 MLST与核心基因组MLST(cgMLST)分析
基于PubMLST(public databases for molecular typing and microbial genome diversity, https://pubmlst.org/)公共数据库获取AB的ST型,采用Phyloviz软件的goeBURST Full MST算法绘制最小生成树;应用基因组流行病学中心在线网站(Center for Genomic Epidemiology, https://www.genomicepidemiology.org/services/)分析cgMLST,并通过iTOL网站(Interactive Tree Of Life https://itol.embl.de/)绘制进化树。
1.5 统计分析
应用SPSS 27.0统计软件对数据进行统计比较,率的组间分析采用卡方检验,以P≤0.05为差异有统计学意义。
2. 结果
2.1 新冠感染大流行前后ICU环境中常见病原菌检出情况
根据上海市及浦东新区耐药菌监测方案,大流行前后共采集3 336份ICU环境标本,监测AB、肺炎克雷伯菌、金黄色葡萄球菌和铜绿假单胞菌这4种常见病原菌,总检出率为8.45%(282株),大流行前病原菌检出率为10.77%(181/1 680),大流行后病原菌检出率为6.10%(101/1 656),大流行前后病原菌检出率比较,差异具有统计学意义(χ2=23.550,P < 0.05)。
4种病原菌中,AB检出率最高,为5.07%(169株),肺炎克雷伯菌、金黄色葡萄球菌、铜绿假单胞菌检出率依次为1.68%(56株)、1.38%(46株)、0.33% (11株),大流行前后AB和金黄色葡萄球菌的检出率比较,差异均具有统计学意义(均P < 0.01),见表 1。
表 1 新冠大流行前后上海市部分医疗机构ICU环境中常见病原菌检出情况Table 1 Detection of common pathogens from the ICU environment of partial medical institutions in Shanghai before and after COVID-19 pandemic菌株 大流行前(n=1 680) 大流行后(n=1 656) χ2 P 检出株数 检出率(%) 检出株数 检出率(%) AB 114 6.79 55 3.32 20.813 < 0.01 金黄色葡萄球菌 38 2.26 8 0.48 19.405 < 0.01 肺炎克雷伯菌 23 1.37 33 1.99 1.966 0.16 铜绿假单胞菌 6 0.36 5 0.30 0.077 0.78 注:n表示采集标本数。 2.2 新冠感染大流行前后ICU环境中检出AB同源性分析
2.2.1 AB MLST分析
对169株AB进行核酸提取、文库构建及测序,并对下机数据进行质控、组装,其中7株菌株因菌株复苏后活力下降,无法提取足量的核酸导致测序失败,共获得162株菌株的全基因组序列,测序成功率为95.86%。
大流行前后,被褥表面AB检出率均为最高。基于巴斯德方案对162株AB全基因组序列进行MLST分析显示,大流行前被褥表面菌株均为ST2型,大流行后被褥表面菌株ST型分布呈多样化,且不同来源AB的ST_Pasteur型分布略有不同,2株新型ST_Pasteur型分别自大流行前工作人员手和洗手液(消毒剂)瓶表面检出,ST164分布于大流行前约束带表面、大流行后护士台面和大流行后便器表面,见表 2。
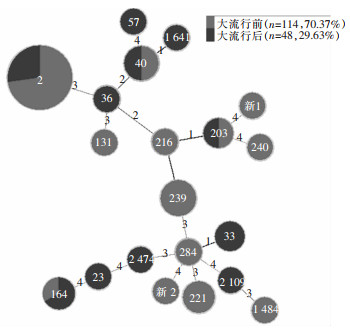
表 2 不同来源AB ST_Pasteur型分布Table 2 Distribution of AB ST-Pasteur types from different sources菌株来源 大流行前 大流行后* 检出株数 医疗机构 ST_Pasteur型 检出株数 医疗机构 ST_Pasteur型 被褥 14 A、B、C、D ST2 14 A、B、C、D、E、F、G、H ST2、ST23、ST36、ST57 床(扶手、床栏等) 10 A、B、C、E ST2、ST40 7 A、D、E、F、G ST2、ST33 约束带 13 A、C、E ST2、ST40、ST164 / / / 工作人员手 8 A、B、C、D、G ST2、ST131、ST221、STnew2 3 H ST40、ST2474 垃圾桶 9 A、B、C、D、E ST2、ST216、ST221 0 0 0 空调进/出风口 7 A、B、C、D、F ST2、ST203、ST221、ST284 / / / 呼吸机 6 A、C ST2 2 B、G ST2 病号服 6 B、C、F ST2、ST1484 / / / 护士台面 5 A、B、F ST2、ST239 1 A ST164 病历夹 4 A、B、E、F ST2、ST239 1 B ST2 工作服 3 A、E ST2 1 E ST2 输液泵 4 C、F ST2、ST239 2 C、E ST2、ST203 洗手液(消毒剂)瓶 3 A、B、C ST2、STnew1 3 A、E、F ST2 枕头 3 A、C、D ST2 0 0 0 门把手 3 B、F ST2、ST239 0 0 0 清洗消毒车 2 A、G ST2、ST239 1 A ST2 水龙头 3 C、F ST2、ST240 1 A ST33 治疗推车 3 A、E、G ST2 2 A、E ST2 心电监护仪 2 C ST2 1 A ST2 键盘 2 A、D ST2 1 A ST2 血压计 2 A ST2 0 0 0 鼻饲管 1 A ST2 0 0 0 电话机 1 C ST2 0 0 0 便器 0 0 0 4 E、G、H ST2、ST164 清洁水桶 0 0 0 1 A ST1641 拖把 0 0 0 3 A、E ST2、ST2109 注:*表示有7株菌测序失败;/表示未采样。 162株AB共检出20种ST_Pasteur型,前三位依次为ST2、ST239、ST40,占比分别为80.25%(130株)、3.09%(5株)、2.47%(4株);2株菌株由于新的管家基因组合无法分型;大流行前后存在4种相同的ST型,分别是ST2、ST40、ST164和ST203,其余ST型均单独出现在大流行前或大流行后。见图 1。
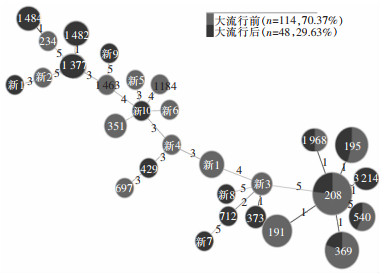
基于牛津方案对162株AB基因组序列进行MLST分析,共获得19种ST型,前三位依次为ST208、ST369、ST195,占比分别为37.04%(60株)、15.43%(25株)、13.58%(22株);11株菌株由于新的管家基因组合无法分型;大流行前后存在5种相同的ST型,分别是ST195、ST208、ST369、ST540和ST1968,其余ST型均单独出现在大流行前或大流行后。见图 2。
2.2.2 基于cgMLST的AB同源性分析
结果显示,162株AB归属于2大分支及多个小分支,各医疗机构的AB存在相同克隆,来自同一所医院的近亲分离株倾向于聚集在同一进化支中。cgMLST聚类将携带ST2_Pasteur的菌株细分超过10个小分支,基于cgMLST和MLST_Pasteur绘制系统发育树,结果显示ST2在遗传上不同于其他序列类型。ST2_Pasteur对应牛津方案的ST208、ST369、ST540、ST3214、ST1968、ST195、ST369和ST191。牛津方案中检出的11种新ST型有10种已在巴斯德方案中被赋予ST型,如大流行前检出的5株ST新1_Oxford菌株与ST239_Pasteur同源,大流行前后的流行克隆仍以ST2_Pasteur/ST208_Oxford为主,但部分等位基因发生了改变,大流行后ST208_Oxford与ST540_Oxford和ST369_Oxford的亲缘关系更接近,ST164_Pasteur克隆由大流行前的ST234_Oxford转变为大流行后的ST1418_Oxford。
3. 讨论
本研究结果显示,大流行前后上海市部分医疗机构ICU环境中病原菌总检出率明显下降,可能与大流行期间消毒措施的优化与加强(加强环境消毒监测与仪器消毒管理等)、工作人员及患者感染防控意识的提高(严格执行手卫生、注重个人卫生与隔离等)、诊疗方式的改变(对患者有序的分流和分区诊疗等)等因素有关,这些干预措施可降低临床环境中病原菌的检出率及耐药细菌的传播[13-16],提示今后仍可继续通过精细化医院感染管理、强化环境消毒和人员隔离等措施减少病原菌的产生及传播。AB是全世界医院感染暴发及ICU细菌感染的首要病原体[16]。本研究中AB的检出率居于首位,患者高频接触的被褥表面和床(扶手、床栏等)表面占比最高,且多所医院的被褥和床(扶手、床栏等)表面均有检出,与文献[16-17]一致,提示应强化对患者高频接触物体表面环境的消毒与监测;大流行后拖把、便器表面的AB检出率高于大流行前,考虑与大流行后加强消毒措施导致拖把等保洁用品使用频率增加有关。
AB主要通过脉冲场凝胶电泳(pulsed-field gel electrophoresis, PFGE)、MALDI-TOF MS和MLST方法进行同源性分析。文献[18-20]表明MLST的分辨特异性明显高于MALDI-TOF MS,正逐渐取代PFGE成为同源性分析的金标准。本研究结果显示,大流行前后AB的流行克隆均以ST2_Pasteur/ST208_Oxford为主,与我国及全球最主要的流行克隆ST2一致,通常与多药耐药和高毒力有关[9, 21],且ST2在不同医疗机构间的密切遗传亲缘性和广泛分布提示可能存在跨医院交叉传播现象。对菌株分离来源、医疗机构和ST_Pasteur联合分析结果显示,大流行前后部分来源菌株的ST型分布发生了明显变化,例如,大流行前被褥表面菌株均为ST2型,大流行后被褥表面菌株的ST型为ST2、ST23、ST36和ST57,可能因大流行期间ICU住院患者流动性增加,消毒和清洁措施对不同ST型菌株消毒效果不同等因素导致患者高频接触被褥物体表面ST型多样化。值得注意的是,ST164菌株可通过产生黑脓素增强对紫外线的抵抗力,具有强大的基因组可塑性和环境适应性[10],本研究中,约束带表面、护士台面和便器表面均检出ST164菌株,需引起医院相关部门的高度重视。
随着全基因组测序的不断进步和公开可用的AB基因组数量的迅速增加,2017年由2 390个基因组成的cgMLST分型方案首次公开,被认为是流行病学研究中最适合和最全面的AB基因分型方法[22-23]。本研究基于cgMLST和2种不同的MLST分型方案的聚类分析结果标明,流行克隆ST2_Pasteur在牛津方案中被进一步区分为ST208、ST369、ST540、ST3214、ST1968、ST195、ST369和ST191,提示牛津方案对近亲分离株的区分能力优于巴斯德方案;另一方面,牛津方案中新发现的11种ST新型有10种属于巴斯德方案中已存在的ST型,提示巴斯德方案更适合在克隆群内进行精确分类,而cgMLST具有更高的辨别力。3者联合分析发现,大流行后的流行克隆虽然仍以ST2_Pasteur/ST208_Oxford为主,但大流行后ST208_Oxford与ST540_Oxford和ST369_Oxford的亲缘关系更接近,且ICU内适应性更强的ST164_Pasteur克隆由大流行前的ST234_Oxford转变为大流行后的ST1418_Oxford,管家基因gpi_Oxford产生了变化。文献[24]表明ST164_Pasteur/ST1418_Oxford可同时携带B类碳青霉烯酶NDM和D类碳青霉烯酶OXA,耐药性更强,需引起高度重视,后续也会结合药敏试验等进一步分析,而这些变化是否与大流行期间消毒剂、抗菌药物使用有关仍需进一步研究。
综上所述,本研究通过分析新冠感染大流行前后ICU环境中常见病原菌的检出率变化来探讨大流行期间精细化感染管理措施的影响,为今后制定消毒等感染控制策略提供数据参考;以AB为研究对象分析大流行前后不同来源菌株同源性变化,发现大流行前后菌株ST型分布存在较大不同,而ST型的变化对消毒效果、患者感染风险等是否存在影响仍需持续监测并进一步探究;对比cgMLST、MLST_Pasteur和MLST_Oxford三种同源性分析方法提示cgMLST在同源性分析、进化与传播、暴发分析方面更具优势,为今后AB的溯源分析提供思路;新发现2种新ST_Pasteur型和11种新ST_Oxford型,丰富了PubMLST数据库,后续也将结合药敏试验等进行深入研究。
利益冲突:所有作者均声明不存在利益冲突。
-
表 1 新冠大流行前后上海市部分医疗机构ICU环境中常见病原菌检出情况
Table 1 Detection of common pathogens from the ICU environment of partial medical institutions in Shanghai before and after COVID-19 pandemic
菌株 大流行前(n=1 680) 大流行后(n=1 656) χ2 P 检出株数 检出率(%) 检出株数 检出率(%) AB 114 6.79 55 3.32 20.813 < 0.01 金黄色葡萄球菌 38 2.26 8 0.48 19.405 < 0.01 肺炎克雷伯菌 23 1.37 33 1.99 1.966 0.16 铜绿假单胞菌 6 0.36 5 0.30 0.077 0.78 注:n表示采集标本数。 表 2 不同来源AB ST_Pasteur型分布
Table 2 Distribution of AB ST-Pasteur types from different sources
菌株来源 大流行前 大流行后* 检出株数 医疗机构 ST_Pasteur型 检出株数 医疗机构 ST_Pasteur型 被褥 14 A、B、C、D ST2 14 A、B、C、D、E、F、G、H ST2、ST23、ST36、ST57 床(扶手、床栏等) 10 A、B、C、E ST2、ST40 7 A、D、E、F、G ST2、ST33 约束带 13 A、C、E ST2、ST40、ST164 / / / 工作人员手 8 A、B、C、D、G ST2、ST131、ST221、STnew2 3 H ST40、ST2474 垃圾桶 9 A、B、C、D、E ST2、ST216、ST221 0 0 0 空调进/出风口 7 A、B、C、D、F ST2、ST203、ST221、ST284 / / / 呼吸机 6 A、C ST2 2 B、G ST2 病号服 6 B、C、F ST2、ST1484 / / / 护士台面 5 A、B、F ST2、ST239 1 A ST164 病历夹 4 A、B、E、F ST2、ST239 1 B ST2 工作服 3 A、E ST2 1 E ST2 输液泵 4 C、F ST2、ST239 2 C、E ST2、ST203 洗手液(消毒剂)瓶 3 A、B、C ST2、STnew1 3 A、E、F ST2 枕头 3 A、C、D ST2 0 0 0 门把手 3 B、F ST2、ST239 0 0 0 清洗消毒车 2 A、G ST2、ST239 1 A ST2 水龙头 3 C、F ST2、ST240 1 A ST33 治疗推车 3 A、E、G ST2 2 A、E ST2 心电监护仪 2 C ST2 1 A ST2 键盘 2 A、D ST2 1 A ST2 血压计 2 A ST2 0 0 0 鼻饲管 1 A ST2 0 0 0 电话机 1 C ST2 0 0 0 便器 0 0 0 4 E、G、H ST2、ST164 清洁水桶 0 0 0 1 A ST1641 拖把 0 0 0 3 A、E ST2、ST2109 注:*表示有7株菌测序失败;/表示未采样。 -
[1] O'Toole RF. The interface between COVID-19 and bacterial healthcare-associated infections[J]. Clin Microbiol Infect, 2021, 27(12): 1772-1776. doi: 10.1016/j.cmi.2021.06.001 [2] 谈宜斌, 王莹, 马晶, 等. 新型冠状病毒感染疫情期间某医院重症监护病房医院感染目标性监测[J]. 中国消毒学杂志, 2023, 40(3): 188-190, 193. Tan YB, Wang Y, Ma J, et al. Targeted surveillance on nosocomial infection in the ICU patients during the COVID-19 epidemic[J]. Chinese Journal of Disinfection, 2023, 40(3): 188-190, 193. [3] Hao L, Yang X, Chen HL, et al. Distribution and drug resistance of bacterial infection in hospitalized patients at the respiratory department before and after the COVID-19 pandemic in Guangzhou, China[J]. Microorganisms, 2023, 11(10): 2542. doi: 10.3390/microorganisms11102542 [4] Nebreda-Mayoral T, Miguel-Gómez MA, March-Rosselló GA, et al. Bacterial/fungal infection in hospitalized patients with COVID-19 in a tertiary hospital in the Community of Castilla y León, Spain[J]. Enferm Infecc Microbiol Clin (Engl Ed), 2022, 40(4): 158-165. doi: 10.1016/j.eimc.2020.11.003 [5] Shinohara DR, Dos Santos Saalfeld SM, Martinez HV, et al. Outbreak of endemic carbapenem-resistant Acinetobacter baumannii in a coronavirus disease 2019 (COVID-19)-specific intensive care unit[J]. Infect Control Hosp Epidemiol, 2022, 43(6): 815-817. doi: 10.1017/ice.2021.98 [6] Durán-Manuel EM, Cruz-Cruz C, Ibáñez-Cervantes G, et al. Clonal dispersion of Acinetobacter baumannii in an intensive care unit designed to patients COVID-19[J]. J Infect Dev Ctries, 2021, 15(1): 58-68. doi: 10.3855/jidc.13545 [7] Hu HB, Lou YF, Feng HT, et al. Molecular characterization of carbapenem-resistant Acinetobacter baumannii isolates among intensive care unit patients and environment[J]. Infect Drug Resist, 2022, 15: 1821-1829. doi: 10.2147/IDR.S349895 [8] Gaiarsa S, Batisti Biffignandi G, Esposito EP, et al. Comparative analysis of the two Acinetobacter baumannii multilocus sequence typing (MLST) schemes[J]. Front Microbiol, 2019, 10: 930. doi: 10.3389/fmicb.2019.00930 [9] Liu CC, Chen KC, Wu YC, et al. Epidemiological and genetic characteristics of clinical carbapenem-resistant Acinetobacter baumannii strains collected countrywide from hospital intensive care units (ICUs) in China[J]. Emerg Microbes Infect, 2022, 11(1): 1730-1741. doi: 10.1080/22221751.2022.2093134 [10] Zhao F, Liu HY, Yao Y, et al. Description of a rare pyomelanin-producing carbapenem-resistant Acinetobacter baumannii strain coharboring chromosomal OXA-23 and NDM-1[J]. Microbiol Spectr, 2022, 10(4): e0214422. doi: 10.1128/spectrum.02144-22 [11] 胡艳芳, 王丹虹, 徐璐艳. 精细化管理在新型冠状病毒疫情期间医院感染防控中的应用[J]. 现代实用医学, 2021, 33(7): 948-949. Hu YF, Wang DH, Xu LY. Application of refined management in prevention and control of nosocomial infection during the novel coronavirus epidemic[J]. Modern Practical Medicine, 2021, 33(7): 948-949. [12] 王振玲, 赖晓全, 徐敏, 等. 某院新型冠状病毒肺炎疫情常态化防控阶段医院感染量化考核方法及效果[J]. 中华医院感染学杂志, 2021, 31(19): 2891-2895. Wang ZL, Lai XQ, Xu M, et al. Effect of quantitative assessment of nosocomial infection management in normalized prevention and control phase of the COVID-19 pneumonia epidemic[J]. Chinese Journal of Nosocomiology, 2021, 31(19): 2891-2895. [13] 章艳菊, 周小娣, 夏云飞. 新型冠状病毒感染疫情前后非定点医院医院感染调查分析[J]. 中国消毒学杂志, 2023, 40(10): 787-790. Zhang YJ, Zhou XD, Xia YF. Investigation and analysis of nosocomial infection in non-designated hospitals before and after the outbreak of novel coronavirus infection[J]. Chinese Journal of Disinfection, 2023, 40(10): 787-790. [14] Rehman S. A parallel and silent emerging pandemic: antimicrobial resistance (AMR) amid COVID-19 pandemic[J]. J Infect Public Health, 2023, 16(4): 611-617. doi: 10.1016/j.jiph.2023.02.021 [15] 张海娇, 杨春艳, 张诤, 等. 基于依从性监测的加强干预对鲍曼不动杆菌感染的防控效果[J]. 中国感染控制杂志, 2022, 21(1): 49-54. https://www.zggrkz.com/zggrkzzz/article/abstract/2022-1-49?st=search Zhang HJ, Yang CY, Zhang Z, et al. Effect of intensive intervention based on compliance monitoring on prevention and control of Acinetobacter baumannii infection[J]. Chinese Journal of Infection Control, 2022, 21(1): 49-54. https://www.zggrkz.com/zggrkzzz/article/abstract/2022-1-49?st=search [16] 切措塔, 何定英, 龙芙艳, 等. 藏区重症监护病房首例耐碳青霉烯类鲍曼不动杆菌感染患者相关环境污染及患者感染情况[J]. 中国感染控制杂志, 2024, 23(2): 220-224. https://www.zggrkz.com/zggrkzzz/article/abstract/2024-2-220?st=search Qie CT, He DY, Long FY, et al. Environmental contamination related to the first patient with carbapenem-resistant Acinetobacter baumannii infection and the infection status of patients in the intensive care unit in Tibetan areas[J]. Chinese Journal of Infection Control, 2024, 23(2): 220-224. https://www.zggrkz.com/zggrkzzz/article/abstract/2024-2-220?st=search [17] 汪要望, 吴安华, 黄勋, 等. 某院综合ICU耐碳青霉烯革兰阴性杆菌的环境流行调查[J]. 中国抗生素杂志, 2022, 47(12): 1305-1311. Wang YW, Wu AH, Huang X, et al. Survey on environmental prevalence of carbapenem-resistant organism in a general ICU[J]. Chinese Journal of Antibiotics, 2022, 47(12): 1305-1311. [18] Shelenkov A, Akimkin V, Mikhaylova Y. International clones of high risk of Acinetobacter baumannii-definitions, history, properties and perspectives[J]. Microorganisms, 2023, 11(8): 2115. [19] 郑恬, 周柯, 周磊, 等. MALDI-TOF MS在鲍曼不动杆菌同源性分析中的临床应用评估[J]. 中国感染控制杂志, 2024, 23(1): 104-111. https://www.zggrkz.com/zggrkzzz/article/abstract/2024-1-104?st=search Zheng T, Zhou K, Zhou L, et al. Clinical application of MALDI-TOF MS for homology analysis of Acinetobacter baumannii[J]. Chinese Journal of Infection Control, 2024, 23(1): 104-111. https://www.zggrkz.com/zggrkzzz/article/abstract/2024-1-104?st=search [20] Wareth G, Linde J, Nguyen NH, et al. WGS-based analysis of carbapenem-resistant Acinetobacter baumannii in Vietnam and molecular characterization of antimicrobial determinants and MLST in Southeast Asia[J]. Antibiotics (Basel), 2021, 10(5): 563. [21] Duan ZM, Li XM, Li S, et al. Nosocomial surveillance of multidrug-resistant Acinetobacter baumannii: a genomic epidemiological study[J]. Microbiol Spectr, 2024, 12(2): e0220723. [22] Higgins PG, Prior K, Harmsen D, et al. Development and evaluation of a core genome multilocus typing scheme for whole-genome sequence-based typing of Acinetobacter baumannii[J]. PLoS One, 2017, 12(6): e0179228. [23] Li TT, Yang YX, Yan RS, et al. Comparing core-genome MLST with PFGE and MLST for cluster analysis of carbapenem-resistant Acinetobacter baumannii[J]. J Glob Antimicrob Resist, 2022, 30: 148-151. [24] Zafer MM, Hussein AFA, Al-Agamy MH, et al. Genomic characterization of extensively drug-resistant NDM-producing Acinetobacter baumannii clinical isolates with the emergence of novel bla ADC-257[J]. Front Microbiol, 2021, 12: 736982.

 下载:
下载: