2. 大理大学基础医学院, 云南 大理 671000
2. School of Basic Medical Sciences, Dali University, Dali 671000, China
严重急性呼吸综合征冠状病毒2(SARS-CoV-2)具有高度传染性,已造成全球社会和经济的混乱。SARS-CoV-2是单正链RNA病毒,大小约为30 kb,属于β冠状病毒属,含至少15个开放阅读框(open reading frames, ORFs),编码4个结构蛋白和16个非结构蛋白。非结构蛋白(non-structural proteins, NSP)包含由ORF1ab编码的NSP1~16和至少9个辅助蛋白ORF3a、3b、6、7a、7b、8b、9b、9c和10[1]。在严重急性呼吸综合征冠状病毒(SARS-CoV)中,结构蛋白之间的相互作用是其成熟所必需,而非结构蛋白之间的结合保证了病毒复制[2]的完成。辅助蛋白在体内和体外病毒复制中必不可少,对病毒-宿主相互作用的其他方面也很重要。
开放阅读框3a(open reading frame 3a,ORF3a) 是冠状病毒最大的辅助蛋白,ORF3a蛋白质由亚基因组RNA3表达,其中包含3a和3b ORFs[3],为复制和毒力所必需,属于SARS-CoV-2基因编码的非结构蛋白。SARS-CoV-2 ORF3a中Q57H突变的发生率超过50%[4],被认为是冠状病毒组装的关键蛋白和细胞毒性蛋白。这些蛋白增强病毒致病性,并在病毒清除后导致神经系统后遗症。开发出特异、安全的候选疫苗对预防SARS-CoV-2感染非常重要。本研究利用现有生物信息学工具评估ORF3a,为研究其免疫原性表位和疫苗设计提供基础。
1 数据与方法 1.1 数据来源使用UniProt数据库分析ORF3a氨基酸序列。
1.2 ORF3a蛋白质的生物信息学分析 1.2.1 理化性质应用在线软件Expasy (https://web.expasy.org/contact)的ProtParam tool对ORF3a蛋白质的理化性质进行预测。
1.2.2 蛋白质结构分析SOPMA和SWISS-MODEL分析ORF3a蛋白质的二、三级结构。
1.2.3 亲疏水性预测通过PortScale分析ORF3a蛋白质的亲疏水性。
1.2.4 跨膜区和信号肽预测应用TMHMM Server 2.0和Signal IP 4.0 Server对ORF3a蛋白质进行跨膜区和信号肽预测。
1.2.5 磷酸化位点分析应用NetPhos 3.1 Server对ORF3a蛋白质的磷酸化进行评分。
1.2.6 T、B细胞抗原表位预测应用ABCpred、SYFPEITHI预测ORF3a蛋白质的B、T细胞抗原表位。
1.2.7 亚细胞定位预测通过ProtComp工具对ORF3a蛋白质亚细胞定位进行分析。
1.2.8 过敏原性、抗原性预测应用VaxiJenv 2.0和AlgPred server预测抗原性和过敏原性。
2 结果 2.1 基因及编码蛋白质基本信息使用蛋白数据库UniProt分析显示,SARS-CoV-2 ORF3a蛋白质由基因3a编码,共275个氨基酸。
2.2 ORF3a蛋白质序列及其理化性质该蛋白质分子量31.123 KDa,等电点5.55,分子式C1440H2189N343O404S11。t1/2=30 h(哺乳动物网织红细胞,体外),t1/2>20 h(酵母,体内),t1/2>10 h(大肠埃希菌,体内)。不稳定指数(instability index, Ⅱ)为32.96,提示为稳定蛋白质(Ⅱ<40为稳定蛋白质,Ⅱ≥40为不稳定蛋白质)。脂肪指数103.42,亲水指数总平均值0.275。ORF3a蛋白质的氨基酸总数为275个,亮氨酸(Leu)占比最多,为10.9%,其次为缬氨酸(Val),占9.1%。
2.3 ORF3a蛋白质结构ORF3a蛋白质的二级结构中无规则卷曲(Cc)93个(占33.82%)。见图 1。三级结构模型见图 2,SWISS-MODEL对模型GMQE评分为0.64,QMEAN评分为0.78±0.05,一致性较好。
 |
| 图 1 ORF3a蛋白质二级结构对应的氨基酸位置 Figure 1 Amino acid position corresponding to secondary structure of ORF3a |
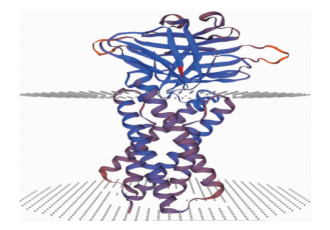 |
| 图 2 ORF3a蛋白质的三级结构预测 Figure 2 Prediction of the tertiary structure of ORF3a protein |
应用ProtScale预测ORF3a为疏水性蛋白质,氨基酸残基分布见图 3。氨基酸亲水性得分为-1.800~3.100(标度值>0的区域较<0的区域密集),总平均亲水指数(GRAVY)为0.275(>0),故预测该蛋白质为疏水性蛋白质。
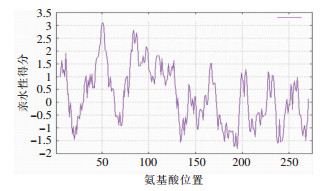 |
| 图 3 ORF3a蛋白质的亲疏水性预测 Figure 3 Prediction of hydrophobicity of ORF3a protein |
ORF3a蛋白质跨膜螺旋数为3。见图 4。跨膜氨基酸数为66.207(>18表示很可能为跨膜蛋白质),在胞质内的概率为0.006,说明ORF3a蛋白质存在跨膜区,为跨膜蛋白质。应用Signal IP 4.0 Server预测ORF3a蛋白质共275个氨基酸,D值为0.181,阈值0.500, 无信号肽。见图 5。
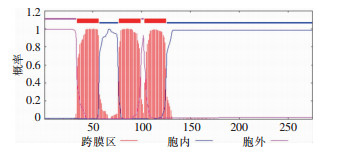 |
| 图 4 ORF3a蛋白质跨膜区预测 Figure 4 Prediction of transmembrane region of ORF3a protein |
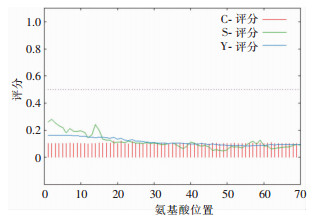 |
| 图 5 ORF3a蛋白质的信号肽预测 Figure 5 Prediction of signal peptide of ORF3a protein |
ORF3a蛋白质含29个磷酸化位点,其中丝氨酸磷酸化位点10个(氨基酸位置为74、92、135、171、177、180、195、209、220、253),苏氨酸磷酸化位点12个(氨基酸位置为9、12、14、24、64、170、190、208、223、269、270、271),酪氨酸磷酸化位点7个(氨基酸位置为156、160、184、189、211、212、264)。见图 6。
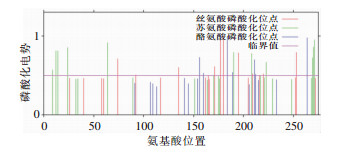 |
| 图 6 ORF3a蛋白质磷酸化位点预测 Figure 6 Prediction of phosphorylation site of ORF3a protein |
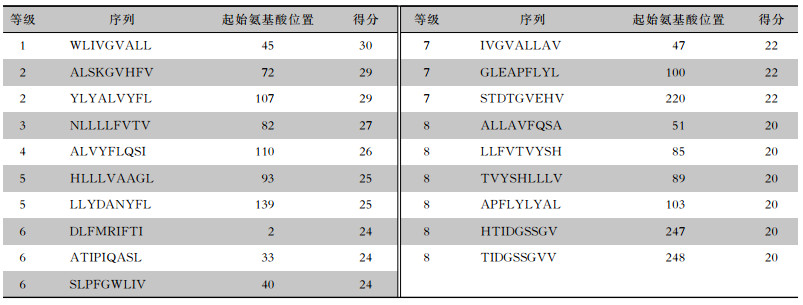
ORF3a蛋白质有多个B细胞抗原表位,其中得分>0.51(临界参考值0.51)的可能B细胞抗原表位24个,>0.85的9个。见表 1。应用SYFPEITHI软件预测获得多个可能的T细胞抗原表位,MHC类型为HLA-A×02 ∶01。见表 2。
| 表 1 ORF3a蛋白B细胞抗原表位预测 Table 1 Prediction of B cell epitopes of ORF3a protein |

|
| 表 2 ORF3a蛋白质T细胞抗原表位预测 Table 2 Prediction of T cell epitopes of ORF3a protein |
|
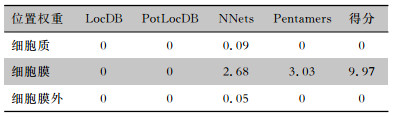
应用ProtComp Version分析该蛋白质最可能定位在宿主细胞的细胞膜,不可能存在于细胞质或细胞膜外。见表 3。
| 表 3 ORF3a蛋白质的亚细胞定位 Table 3 Subcellular localization of ORF3a protein |
|
基于蛋白质的主要化学特征,应用VaxiJenv 2.0预测其抗原性,使用针对各种目标生物体(细菌、病毒、肿瘤、寄生虫和真菌)的非对齐方法,准确率为70%~89%。本研究选择“病毒”为目标生物体(阈值0.4),总体预测抗原可能性为0.4945,具有抗原性。应用AlgPred server预测ORF3a蛋白质为非过敏原性,不诱导过敏原特异性抗体的产生。
3 讨论SARS-CoV-2感染是21世纪第三次传染病大流行,病毒传播速度快、生存能力强、致死率高,已致全球600多万人丧失生命。虽然目前疫苗在预防SARS-CoV-2感染方面有效,但新的病毒突变体可能会削弱现有疫苗的有效性,所以迫切需要找出突变体相关疫苗,推进SARS-CoV-2感染预防方案的合理设计。SARS-CoV-2是一种高度复杂和多样化的RNA病毒,具有大基因组、复杂的基因库,以及转录和翻译机制,感染后可引起高发病率和高病死率的严重呼吸道疾病。与其他冠状病毒一样,SARS-CoV-2独特的辅助蛋白可以破坏人体先天免疫机制,可能与病毒高致病性有关,并在病毒复制中发挥重要作用。因此,研究这些蛋白质可能阐明SARS-CoV-2破坏细胞功能的致病机制。近年来,生物信息学以及新的计算和预测工具在现代结构疫苗学中的应用极大地促进了新型疫苗的开发。在此,通过生物信息学研究展示ORF3a作为SARS-CoV-2疫苗靶点的潜在意义。
ORF3a是SARS-CoV-2最重要的辅助蛋白质之一,也是SARS-CoV-2基因组编码的最大辅助蛋白质。ORF3a区域含有G-四联体结构,该结构在人类和其他生物的基因转录中发挥调节作用,可促进SARS-CoV-2的转录和复制[5]。ORF3a基因位于病毒基因组中的刺突蛋白和包膜蛋白基因之间,其蛋白的主要作用包括毒性、传染性、离子通道活性、形态发生和病毒释放。频率最高的突变是Q57H[6],可能会改变病毒的传播和发病机制,故了解其复杂的突变特征可以指导疫苗的设计和开发。此外,ORF3a蛋白质的主要特征之一是存在一个富含半胱氨酸的结构域,其参与酶的亲核取代反应和蛋白-蛋白相互作用[7]。研究[8]发现,SARS-CoV-2 ORF3a与SARS-CoV ORF3a具有72%的同源性,可形成同型四聚体钾敏感离子通道,诱导细胞周期阻滞和细胞凋亡,并影响病毒的摄取和释放,但SARS-CoV-2 ORF3a的凋亡活性低于SARS-CoV ORF3a,故SARS-CoV-2感染者在早期阶段症状较轻甚至无症状[9]。感染后期,ORF3a激活IL-1β前基因表达和IL-1β分泌,可导致严重的肺损伤[10]。研究[11]通过高通量筛选试验发现,ORF3a蛋白质与人血红素加氧酶1(heme oxygenase 1, HMOX1)蛋白高度相互作用,并抑制HMOX1的抗炎功能,从而加重炎症和组织损伤。此外,ORF3a也能阻止自噬体与溶酶体融合,保护病毒不被破坏[12]。
本研究采用生物信息学方法预测ORF3a蛋白质含有若干T、B细胞抗原表位,具有抗原性和非过敏原性,可用于多表位疫苗的设计,使其有效地识别和组装更能刺激免疫系统的B细胞和T细胞表位,诱导免疫反应。ORF3a蛋白质的不稳定性指数<40,被认为是稳定的蛋白质,故所设计的肽疫苗是稳定的。高脂肪指数(103.42)表明蛋白质在较宽温度范围内具有较高的稳定性。本研究预测ORF3a蛋白质主要位于宿主细胞的细胞膜,与Issa等[8]研究一致。ORF3a也被证明定位于质膜、核周区域和高尔基体[13],并诱导细胞凋亡、病毒颗粒释放、细胞毒性[14]、高尔基体碎片和细胞内囊泡的形成[15]。ORF3a具有3个跨膜螺旋区(TM1-TM3,残基41-132),具有促凋亡功能,目前已分离出17个突变体,其中10个突变位点出现在ORF3a的跨膜结构域内,并与中心孔或离子通道接触。其他变异位点位于ORF3a结构的不同位置,最常见的突变是Q57H。尽管该突变点位于跨膜通道,但通道的几何形状没有发生显著改变,表明通道特性没有改变[16],ORF3a结构域的相对保守性提示了疫苗设计的可能性。研究预测ORF3a是疏水性蛋白质,能够在宿主细胞膜上形成孔,被称为“病毒孔蛋白”。病毒孔蛋白定位于宿主细胞膜结构,如内质网、高尔基体,可以促进离子的运输,调节离子稳态,为病毒感染创造有利环境,也促进病毒免疫逃逸。SARS-CoV-2感染给医疗行业带来重大压力,如何进行有效的预防和治疗成为亟待解决的问题。对SARS-CoV-2的蛋白进行生信分析有助于了解病毒自身结构和侵染过程,根据蛋白的抗原表位进行疫苗研发,使疾病预防更有针对性。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Chan JFW, Kok KH, Zhu Z, et al. Genomic characterization of the 2019 novel human-pathogenic coronavirus isolated from a patient with atypical pneumonia after visiting Wuhan[J]. Emerg Microbes Infect, 2020, 9(1): 221-236. DOI:10.1080/22221751.2020.1719902 |
| [2] |
Snijder EJ, Decroly E, Ziebuhr J. The nonstructural proteins directing coronavirus RNA synthesis and processing[J]. Adv Virus Res, 2016, 96: 59-126. |
| [3] |
Rota PA, Oberste MS, Monroe SS, et al. Characterization of a novel coronavirus associated with severe acute respiratory syndrome[J]. Science, 2003, 300(5624): 1394-1399. DOI:10.1126/science.1085952 |
| [4] |
Wu SQ, Tian C, Liu PP, et al. Effects of SARS-CoV-2 mutations on protein structures and intraviral protein-protein inte-ractions[J]. J Med Virol, 2021, 93(4): 2132-2140. DOI:10.1002/jmv.26597 |
| [5] |
Ji DY, Juhas M, Tsang CM, et al. Discovery of G-quadruplex-forming sequences in SARS-CoV-2[J]. Brief Bioinform, 2021, 22(2): 1150-1160. DOI:10.1093/bib/bbaa114 |
| [6] |
Hassan SS, Attrish D, Ghosh S, et al. Pathogenic perspective of missense mutations of ORF3a protein of SARS-CoV-2[J]. Virus Res, 2021, 300: 198441. DOI:10.1016/j.virusres.2021.198441 |
| [7] |
To J, Torres J. Beyond channel activity: protein-protein intera-ctions involving viroporins[J]. Subcell Biochem, 2018, 88: 329-377. |
| [8] |
Issa E, Merhi G, Panossian B, et al. SARS-CoV-2 and ORF3a: nonsynonymous mutations, functional domains, and viral pathogenesis[J]. mSystems, 2020, 5(3): e00266-20. |
| [9] |
Ren YJ, Shu T, Wu D, et al. The ORF3a protein of SARS-CoV-2 induces apoptosis in cells[J]. Cell Mol Immunol, 2020, 17(8): 881-883. DOI:10.1038/s41423-020-0485-9 |
| [10] |
Siu KL, Yuen KS, Castaño-Rodriguez C, et al. Severe acute respiratory syndrome coronavirus ORF3a protein activates the NLRP3 inflammasome by promoting TRAF3-dependent ubi-quitination of ASC[J]. FASEB J, 2019, 33(8): 8865-8877. DOI:10.1096/fj.201802418R |
| [11] |
Batra N, De Souza C, Batra J, et al. The HMOX1 pathway as a promising target for the treatment and prevention of SARS-CoV-2 of 2019(COVID -19)[J]. Int J Mol Sci, 2020, 21(17): 6412. DOI:10.3390/ijms21176412 |
| [12] |
Miao GY, Zhao HY, Li Y, et al. ORF3a of the COVID -19 virus SARS-CoV-2 blocks HOPS complex-mediated assembly of the SNARE complex required for autolysosome formation[J]. Dev Cell, 2021, 56(4): 427-442.e5. DOI:10.1016/j.devcel.2020.12.010 |
| [13] |
Yuan XL, Li JY, Shan YJ, et al. Subcellular localization and membrane association of SARS-CoV 3a protein[J]. Virus Res, 2005, 109(2): 191-202. DOI:10.1016/j.virusres.2005.01.001 |
| [14] |
Freundt EC, Yu L, Goldsmith CS, et al. The open reading frame 3a protein of severe acute respiratory syndrome-associa-ted coronavirus promotes membrane rearrangement and cell death[J]. J Virol, 2010, 84(2): 1097-1109. DOI:10.1128/JVI.01662-09 |
| [15] |
Yang S, Tian MJ, Dai YL, et al. Infection and chronic disease activate a brain-muscle signaling axis that regulates muscle performance[J]. bioRxiv, 2022. DOI: 10.1101/2020.12.20.423533. Epubaheadofprint.
|
| [16] |
Bianchi M, Borsetti A, Ciccozzi M, et al. SARS-CoV-2 ORF3a: mutability and function[J]. Int J Biol Macromol, 2021, 170: 820-826. DOI:10.1016/j.ijbiomac.2020.12.142 |



