2. 中国人民解放军疾病预防控制中心应急处置大队, 北京 100071;
3. 安徽医科大学公共卫生学院流行病与统计学系, 安徽 合肥 230032;
4. 中国人民解放军疾病预防控制中心有害生物防制科, 北京 100071;
5. 郑州大学公共卫生学院, 河南 郑州 450001;
6. 中国人民解放军疾病预防控制中心信息科, 北京 100071;
7. 中国人民解放军疾病预防控制中心卫勤部, 北京 100071
2. Department of Emergency Response, Chinese PLA Center for Disease Control and Prevention, Beijing 100071, China;
3. Department of Epidemiology and Statistics, School of Public Health, Anhui Medical University, Hefei 230032, China;
4. Department of Pest Management, Chinese PLA Center for Disease Control and Prevention, Beijing 100071, China;
5. College of Public Health, Zhengzhou University, Zhengzhou 450001, China;
6. Department of Information, Chinese PLA Center for Disease Control and Prevention, Beijing 100071, China;
7. Department of Health Service, Chinese PLA Center for Disease Control and Prevention, Beijing 100071, China
肺炎克雷伯菌(Klebsiella pneumoniae,KP)属于条件致病菌,是常见的临床患者感染的致病菌之一。抗菌药物在临床中广泛使用,导致产生耐药的KP数量越来越多。耐碳青霉烯类肺炎克雷伯菌(carbapenem-resistant Klebsiella pneumoniae,CRKP)是临床常见耐药菌,其检出率呈不断升高趋势[1-3]。临床CRKP常见标本来源包括尿、血、呼吸道分泌物和无菌体液等[2]。年老体弱、侵袭性操作[4]和基础疾病[5]是临床患者感染CRKP的主要因素。近年来,CRKP所致血流感染的病例越来越多[6],该类患者病情凶险、预后差,部分文献报道CRKP血流感染患者病死率高达71.9%,远高于其他部位感染的病死率(21.9%)[7],CRKP已成为全球范围内患者正在面临的严重安全威胁。为研究不同标本来源CRKP在携带耐药基因、毒力基因和质粒数量等方面有无差异,笔者对公共数据库2011—2020年的CRKP基因组测序数据进行筛选分析,为深入了解不同标本类型CRKP感染的临床特点和感染控制提供参考。
1 材料与方法 1.1 数据来源和筛选标准本研究从Pathosystems Resource Integration Center (PATRIC)[8]数据库(PATRIC数据库提供全面的细菌基因组学数据和与基因组分析相关的大量数据,相比于NCBI数据库,标本来源、分离国家、耐药情况等基础信息更完善,更便于进行比较分析)下载2011—2020年间所有的KP基因组测序数据,根据宿主、标本来源、对碳青霉烯类抗生素耐药等信息进行筛选,纳入标准包括:(1)人类宿主;(2)按照2020年中国细菌耐药检测结果的分类情况[2]并结合收集到的标本类型,确定标本来源为血、尿、肺泡灌洗液、无菌体液、伤口脓液、粪便、呼吸道标本和导管标本共8个类型;(3)菌株携带碳青霉烯酶基因;(4)分离国家等基础信息完整。排除标准:(1)分离自人类以外的宿主;(2)除血、尿、肺泡灌洗液、无菌体液、伤口脓液、粪便、呼吸道标本和导管标本以外的标本来源;(3)不携带碳青霉烯酶基因;(4)分离时间、分离地区、标本来源、基因组数据等信息不完整。
1.2 毒力基因、耐药基因、质粒和单核苷酸多态性(single nucleotide polymorphisms, SNPs)分析应用kleborate[9]软件对菌株进行物种鉴定,并筛选其是否携带CARD[10]数据库中存在的碳青霉烯酶基因。应用abricate软件,将基因组序列与CARD、VFDB[11]和PlasmidFinder[12]数据库进行比对,获得每个菌株携带毒力基因、耐药基因和质粒的情况。选择肺炎克雷伯菌HS11286 (GenBank assembly accession: GCA_000240185.2)作为参考基因组。应用snippy软件,将各菌株基因组与参考基因组进行对比分析,获得各菌株SNPs数量。
1.3 统计学分析应用IBM SPSS 23.0和R v4.2.0软件进行统计学分析。毒力基因、耐药基因和质粒等计数资料采用中位数[M(P25,P75)]进行统计描述,采用非参数检验(Kruskal-Wallis H秩和检验)比较多个组间总体差异,曼-惠特尼U检验比较两组间差异。采用卡方检验对不同率或百分比之间的差异进行统计学比较,P≤0.05为差异具有统计学意义。
2 结果 2.1 CRKP基因组序列的筛选和标本分布对2011—2020年10年间PATRIC公共数据库中12 774株KP进行筛选后纳入2 356个符合筛选条件的CRKP基因组序列进行分析,其中2011—2015年1 621株(20.92%,共计7 749株),2016—2020年735株(14.63%,共计5 025株)。尿(797株,33.83%)、血(706株,29.97%)、呼吸道(366株,15.53%)和粪便(231株,9.80%)为主要标本来源。血标本CRKP的数量每年均位居前3位,提示血是CRKP最常见标本来源之一。见图 1。
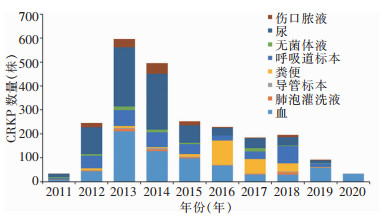 |
| 图 1 2011—2020年不同标本来源CRKP的数量分布 Figure 1 Distribution of CRKP from different specimen sources, 2011-2020 |
美国在2013—2014年提交至数据库的KP感染基因组数据较多,使这两年纳入分析的CRKP基因组样本明显增多,2013年397株,2014年296株。2011—2015年美国提交CRKP基因组序列数量最多(n=1 001),占该5年所有CRKP序列数量的61.75%。2016—2020年提交CRKP基因组序列数量最多的国家分别是泰国(n=188)、中国(n=187)、巴西(n=93)和印度(n=69),占这5年所有CRKP序列数量的73.06%,其他国家/地区提交CRKP基因组序列总体较少。见表 1。
| 表 1 2011—2020年不同国家/地区提交CRKP基因组序列数量(株) Table 1 CRKP genome sequence numbers submitted by different countries/regions, 2011-2020 (No. of strains) |
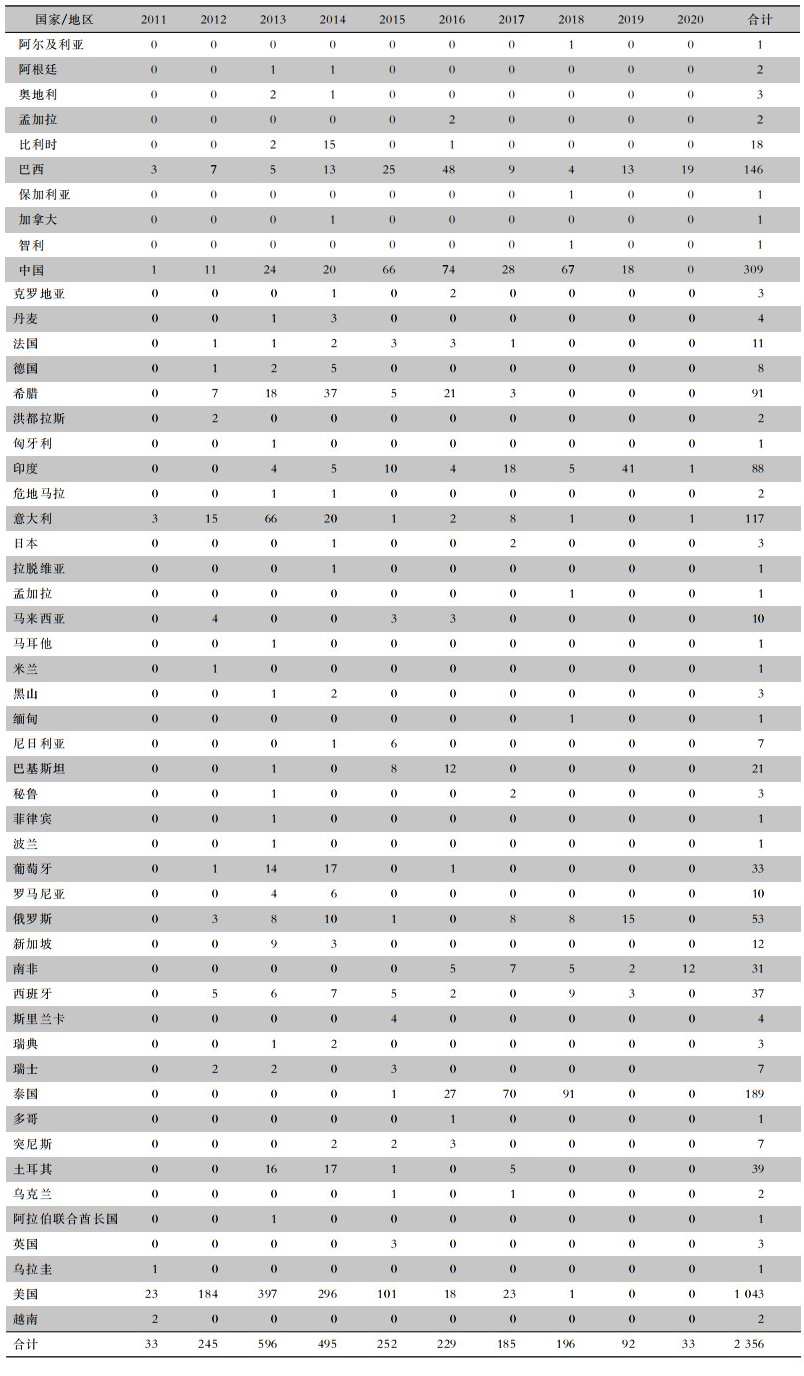
|
2 356株CRKP菌株中最常见的5种碳青霉烯酶基因为:blaKPC-2(50.51%)、blaKPC-3(21.39%)、blaNDM-1(11.50%)、blaOXA-232(10.48%)和blaOXA-48(7.39%)。7.05%的CRKP菌株携带多个碳青霉烯酶基因。美国来源的CRKP菌株携带的碳青霉烯酶以blaKPC-2和blaKPC-3为主,巴西和中国以携带blaKPC-2为主,泰国主要携带blaOXA-232和blaNDM-1,意大利和葡萄牙以blaKPC-3为主,俄国、西班牙主要携带blaOXA-48。产KPC-2(50.51%)是CRKP对碳青霉烯类药物耐药的主要机制,但不同碳青霉烯酶基因在标本整体和不同标本类型中占比不同。blaKPC-2整体占比由2011—2015的54.8%降至2016—2020的41.1%,差异有统计学意义(P < 0.001),除blaKPC-2外的其他碳青霉烯酶基因占比在2016—2020年里均明显增加。尿中以blaKPC-2(46.05%)、blaKPC-3(31.62%)、blaNDM-1(9.91%)和blaOXA-48(7.78%)为主;血中以blaKPC-2(51.98%)、blaKPC-3(22.10%)、blaOXA-232(8.22%)和blaOXA-48(6.94%)为主;呼吸道标本以blaKPC-2(59.56%)、blaOXA-232(20.22%)和blaNDM-1(14.21%)为主;粪便以blaKPC-2(50.22%)、blaOXA-232(36.36%)和blaNDM-1(31.17%)为主;伤口脓液中以blaKPC-2(48.89%)、blaOXA-48(20.74%)和blaKPC-3(18.52%)为主;无菌体液以blaKPC-2(48.39%)、blaKPC-3(27.42%)和blaOXA-48(9.68%)为主;肺泡灌洗液以blaKPC-2(47.37%)、blaKPC-3(26.32%)和blaNDM-1(10.53%)为主;导管标本以blaKPC-2(38.10%)、blaNDM-1和blaOXA-48(均19.05%)为主。见表 2。
| 表 2 2011—2020年不同碳青霉烯酶基因在8种类型标本中的分布情况[株(%)] Table 2 Distribution of different carbapenemase genes among 8 types of specimens, 2011-2020 (No. of isolates [%]) |
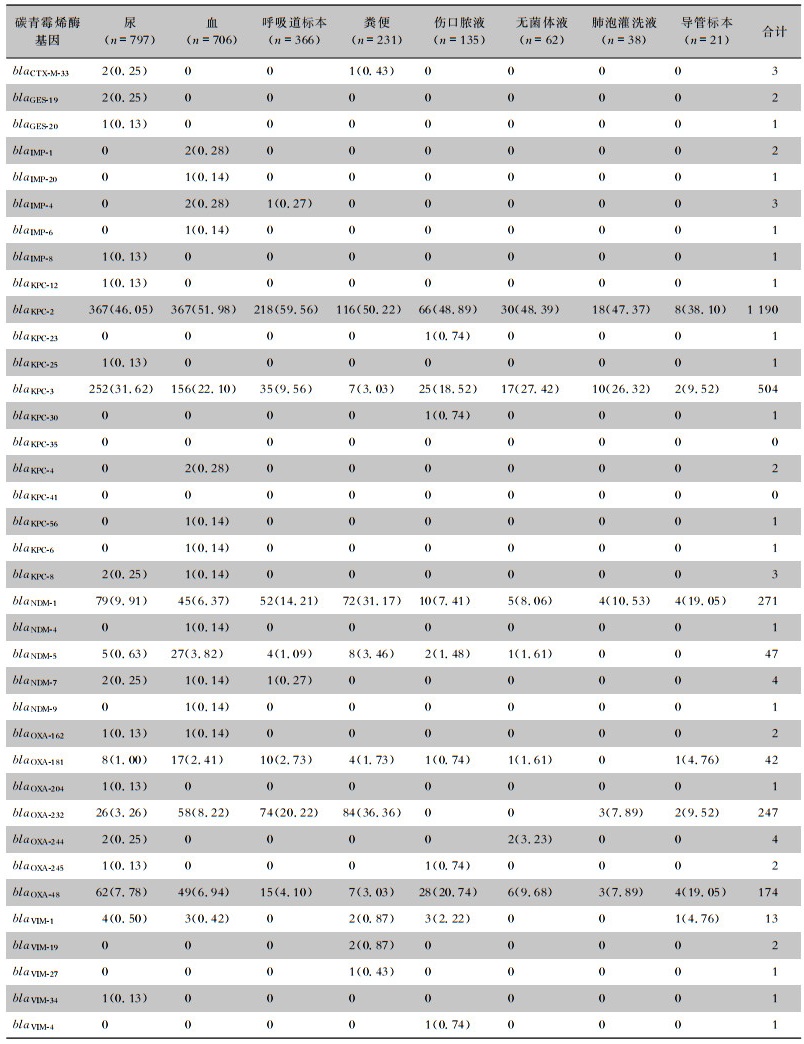
|
2 356株CRKP可划分为133个多位点序列型别(ST型),其中,2011—2015年有101个ST型,2016—2020年有65个ST型。常见的型别包括ST258(33.19%, 782株)、ST11(16.00%, 377株)、ST307(8.91%, 210株)、ST16(7.77%, 183株)、ST512(5.01%, 118株)、ST15(3.82%, 90株)和ST147(3.18%, 75株)。ST258型主要集中在2012—2015年(90.15%,705/782);ST11型2015、2016和2018年最多,占所有ST11型的62.60%(236/377);ST307型2012—2015年出现较多(89.05%,187/210);ST16型CRKP主要出现在2016—2019年(89.07%,163/183)。
2.5 不同标本类型CRKP耐药、质粒和毒力基因携带数量比较CRKP菌株携带耐药基因、质粒和毒力基因数量的M(P25,P75)分别为18(16,20)、4(3,5)和60(57,68)。不同标本来源的CRKP携带耐药基因数量比较,差异无统计学意义(P=0.055),携带毒力基因和质粒数量之间差异均有统计学意义(P < 0.001)。粪便来源的CRKP菌株携带耐药基因、质粒和毒力基因的数量较多,其M(P25,P75)分别为19(16,21)、5(3,6)、63(61,69);导管来源CRKP主要携带iutA、iucA、iucB、iucC、iucD(均为19.0%,4/21);rmpA和rmpA2在肺泡灌洗液来源CRKP中数量最多,分别占比7.9%(3/38)和15.8%(6/38);无菌体液中CRKP携带iro(iroB、iroC、iroD、iroN)基因比率高于其他标本类型CRKP(6.5%,4/62),不同标本类型的CRKP携带关键毒力基因(iutA、iucA、iucB、iucC、iucD、rmpA、rmpA2、iroB、iroC、iroD、iroN)数量差异有统计学意义(P < 0.001)。见表 3。
| 表 3 不同标本来源CRKP耐药基因、质粒和毒力基因携带情况 Table 3 Drug-resistant genes, plasmids and virulence genes carried by CRKP from different specimen sources |
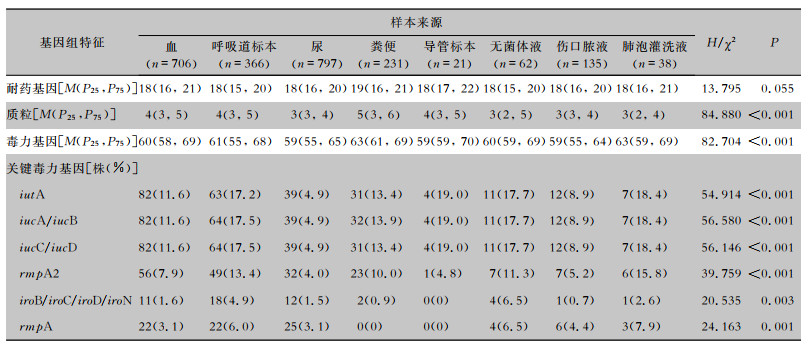
|
不同标本来源的CRKP菌株SNPs数量为2 924~41 470(P=0.414),其中呼吸道分泌物CRKP菌株SNP变异位点数量最少[7 419(7 017, 24 156)],粪便标本CRKP含有SNPs变异位点数量最多[20 148(3 528, 24 233)],不同样本类型CRKP携带SNPs的数量见图 2。
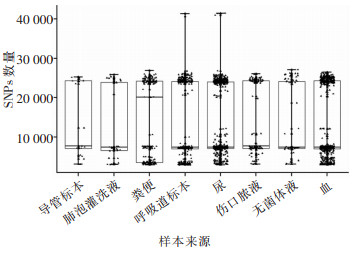 |
| 图 2 不同标本类型CRKP的SNPs数量比较 Figure 2 Comparison of SNPs amount in CRKP from diffe-rent types of specimens |
对706份血标本与1 650份非血标本来源的CRKP耐药基因、毒力基因和质粒携带数量进行比较,发现血标本来源CRKP[60(58, 69)]比其他标本类型CRKP[60(56, 67)]携带更多毒力基因,差异有统计学意义(P=0.004)。见表 4。
| 表 4 血标本与其他标本来源的CRKP耐药基因、质粒、毒力基因和SNPs携带情况 Table 4 Analysis of drug-resistant genes, plasmids, virulence genes and SNPs carried by CRKP from blood specimens and other specimen sources |
|
国内外CRKP的感染情况日益严重,对患者入院治疗和预后影响巨大。以往关于CRKP的研究主要为基于局部地区所有标本类型的研究或单一标本来源的研究,如某医院、某区域所有CRKP菌株的流行病学特征和基因组研究,以探究其流行、传播和进化机制,但比较不同标本类型CRKP差异的相关研究还不充分。
本研究对2011—2020年PATRIC数据库中所有符合筛选标准的CRKP进行分析,发现尿、血和呼吸道标本是临床CRKP感染的主要标本类型。与以往研究[13]相比,血标本CRKP在所有CRKP中的占比有所提升。美国在2013—2014年提交CRKP的数量较多,可能与该时间段美国对KP的关注突然增加有关。美国和中国分别以ST258型(63.66%)和ST11型(79.22%)为主要序列类型,与以往研究[14-15]相同。10年间,不同碳青霉烯酶类型在各标本来源CRKP中的比例发生改变,虽仍以KPC-2为主,但其他类型碳青霉烯酶占比增加,提示CRKP菌株的耐药基因型向复杂多样化进化。
CRKP往往携带多种碳青霉烯酶耐药基因,常见blaKPC-2和blaNDM-1,主要流行于ST11型CRKP菌株;blaVIM-1、blaVIM-2、blaOXA-232主要流行于ST23型CRKP菌株[16]。血标本来源CRKP以产碳青霉烯酶为最常见的耐药机制,编码该酶的基因位于细菌质粒或染色体上,通过转座子或结合性质粒等移动元件在菌株与人群之间广泛传播。各个国家分离的菌株产碳青霉烯酶类型各不相同,与既往研究[17]一致,在美国,产KPC酶为CRKP最常见耐药机制;OXA-48和NDM-1是埃及癌症患者感染的CRKP中最常见的类型[18]。本组研究显示,分离自泰国的CRKP菌株主要携带NDM-1,质粒介导的KPC-2的水平传播是我国临床CRKP的主要耐药机制,俄国、西班牙和葡萄牙等欧洲国家的CRKP菌株主要携带OXA-48。许多地区都存在主要流行的碳青霉烯酶类型,提示地理位置和区域管辖对CRKP的传播进化影响密切。
血标本来源的CRKP在携带质粒和毒力基因(iutA、iucA、iucB、iucC、iucD、rmpA、rmpA2、iroB、iroC、iroD、iroN等)方面与其他7个标本类型存在差异,血标本中分离的CRKP携带更多数量的毒力基因,差异有统计学意义(P=0.004),提示血标本来源CRKP感染者的高病死率可能与菌株携带更多毒力基因有关。既往研究也在所有血标本肺炎克雷伯菌中检测到毒力基因,其中84.13%的菌株携带高毒力基因[19],携带iutA等高毒力基因是血流感染CRKP患者死亡的独立危险因素[20],证实CRKP菌株携带毒力基因与患者感染后高病死率有直接关联。
本研究存在一定局限性,首先,由于PATRIC公共数据库菌株信息受全球地区不同经济状况及提交数据课题组研究方向偏好等因素影响,菌株基因组信息代表性可能不够强,但该数据库提供了较完善的菌株背景信息,为研究肺炎克雷伯菌的基因组特征提供了较好的资源。其次,现有研究显示菌株基因型和表型之间还存在差异,但由于本研究对象均为PATRIC公共数据库的基因组信息,缺乏相关实验研究证实。
本研究通过分析从PATRIC公共数据库中收集的不同标本类型的CRKP携带毒力基因、耐药基因和质粒数量情况,发现不同标本类型的CRKP携带毒力基因和质粒数量差异有统计学意义,血标本CRKP相比其他标本来源的CRKP携带更多的毒力基因,在一定程度上反映了公共数据库中不同标本来源CRKP的分布特征,以及耐药基因、质粒和毒力基因的携带水平,为后续CRKP的耐药和毒力相关研究提供参考。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
全国细菌耐药监测网. 全国细菌耐药监测网2014-2019年耐碳青霉烯类肺炎克雷伯菌流行病学变迁[J]. 中国感染控制杂志, 2021, 20(2): 175-179. China Antimicrobial Resistance Surveillance System. Epidemiological change in carbapenem-resistant Klebsiella pneumoniae: surveillance report from China Antimicrobial Resistance Surveillance in 2014-2019[J]. Chinese Journal of Infection Control, 2021, 20(2): 175-179. |
| [2] |
胡付品, 郭燕, 朱德妹, 等. 2020年CHINET中国细菌耐药监测[J]. 中国感染与化疗杂志, 2021, 21(4): 377-387. Hu FP, Guo Y, Zhu DM, et al. CHINET surveillance of bacterial resistance: results of 2020[J]. Chinese Journal of Infection and Chemotherapy, 2021, 21(4): 377-387. |
| [3] |
姚志宏, 刘真真. 耐碳青霉烯类肺炎克雷伯菌的流行病学、耐药与传播机制研究进展[J]. 中国抗生素杂志, 2017, 42(12): 1107-1112. Yao ZH, Liu ZZ. Advances in epidemiology, drug resistance and transmission mechanism of carbapenem-resistant Klebsiella pneumoniae[J]. Chinese Journal of Antibiotics, 2017, 42(12): 1107-1112. DOI:10.3969/j.issn.1001-8689.2017.12.017 |
| [4] |
詹忠明, 曹敏. 耐碳青霉烯类肺炎克雷伯菌院内感染患者的耐药特点及其相关危险因素分析[J]. 抗感染药学, 2021, 18(7): 983-986. Zhan ZM, Cao M. Characteristics of drug resistance and its associated risk factors in patients with carbapenem-resistant Klebsiella pneumoniae nosocomial infections[J]. Anti-Infection Pharmacy, 2021, 18(7): 983-986. |
| [5] |
杨修文, 崔俊昌, 赵进, 等. 医院获得性肺炎克雷伯菌血流感染的临床特征及耐药菌株感染的危险因素分析[J]. 解放军医学院学报, 2017, 38(11): 1029-1032, 1054. Yang XW, Cui JC, Zhao J, et al. Carbapenem-susceptible versus carbapenem-resistant Klebsiella pneumoniae bloodstream infection: clinical manifestations and risk factors[J]. Academic Journal of Chinese PLA Medical School, 2017, 38(11): 1029-1032, 1054. DOI:10.3969/j.issn.2095-5227.2017.11.008 |
| [6] |
全国细菌耐药监测网. 全国细菌耐药监测网2014-2019年血标本病原菌耐药性变迁[J]. 中国感染控制杂志, 2021, 20(2): 124-133. China Antimicrobial Resistance Surveillance System. Change in antimicrobial resistance of pathogens from blood specimens: surveillance report from China Antimicrobial Resistance Surveillance System in 2014-2019[J]. Chinese Journal of Infection Control, 2021, 20(2): 124-133. |
| [7] |
李媛媛. 五年间血流感染耐碳青霉烯类肺炎克雷伯菌耐药监测及感染、死亡危险因素分析[D]. 石家庄: 河北医科大学, 2020. Li YY. Five-year change of resistance surveillance and risk factors for infection and mortality of carbapenem-resistant Klebsiella pneumoniae bloodstream infection[D]. Shijiazhuang: Hebei Medical University, 2020. |
| [8] |
Gillespie JJ, Wattam AR, Cammer SA, et al. PATRIC: the comprehensive bacterial bioinformatics resource with a focus on human pathogenic species[J]. Infect Immun, 2011, 79(11): 4286-4298. DOI:10.1128/IAI.00207-11 |
| [9] |
Lam MMC, Wick RR, Watts SC, et al. A genomic surveillance framework and genotyping tool for Klebsiella pneumo-niae and its related species complex[J]. Nat Commun, 2021, 12(1): 4188. DOI:10.1038/s41467-021-24448-3 |
| [10] |
Jia BF, Raphenya AR, Alcock B, et al. CARD 2017:expansion and model-centric curation of the comprehensive antibiotic resistance database[J]. Nucleic Acids Res, 2017, 45(D1): D566-D573. DOI:10.1093/nar/gkw1004 |
| [11] |
Chen LH, Zheng DD, Liu B, et al. VFDB 2016:hierarchical and refined dataset for big data analysis-10 years on[J]. Nucleic Acids Res, 2016, 44(D1): D694-D697. DOI:10.1093/nar/gkv1239 |
| [12] |
Carattoli A, Zankari E, García-Fernández A, et al. In silico detection and typing of plasmids using PlasmidFinder and plasmid multilocus sequence typing[J]. Antimicrob Agents Chemother, 2014, 58(7): 3895-3903. |
| [13] |
李雪娇, 马炜, 郭杰, 等. 耐碳青霉烯类肺炎克雷伯菌的流行病学特征及分子生物学研究[J]. 第二军医大学学报, 2020, 41(10): 1109-1114. Li XJ, Ma W, Guo J, et al. Epidemiological characteristics and molecular biology of carbapenem-resistant Klebsiella pneumoniae[J]. Academic Journal of Second Military Medical University, 2020, 41(10): 1109-1114. |
| [14] |
Chen L, Mathema B, Pitout JDD, et al. Epidemic Klebsiella pneumoniae ST258 is a hybrid strain[J]. mBio, 2014, 5(3): e01355-14. |
| [15] |
Dong N, Zhang R, Liu LZ, et al. Genome analysis of clinical multilocus sequence type 11 Klebsiella pneumoniae from China[J]. Microb Genom, 2018, 4(2): e000149. |
| [16] |
郑茂, 邹玉, 刘晓, 等. 耐碳青霉烯类高毒力肺炎克雷伯菌的研究进展[J]. 中国感染与化疗杂志, 2021, 21(1): 105-110. Zheng M, Zou Y, Liu X, et al. Research advances in carbapenem-resistant hypervirulent Klebsiella pneumoniae[J]. Chinese Journal of Infection and Chemotherapy, 2021, 21(1): 105-110. |
| [17] |
Kitchel B, Rasheed JK, Patel JB, et al. Molecular epidemiology of KPC-producing Klebsiella pneumoniae isolates in the United States: clonal expansion of multilocus sequence type 258[J]. Antimicrob Agents Chemother, 2009, 53(8): 3365-3370. |
| [18] |
ElMahallawy HA, Zafer MM, Amin MA, et al. Spread of carbapenem resistant Enterobacteriaceae at tertiary care cancer hospital in Egypt[J]. Infect Dis (Lond), 2018, 50(7): 560-564. |
| [19] |
鞠启文, 王运中, 陶云珍, 等. 儿童血流感染肺炎克雷伯菌毒力与耐药基因分析[J]. 中华医院感染学杂志, 2021, 31(21): 3337-3341. Ju QW, Wang YZ, Tao YZ, et al. Virulence and drug resis-tance genes in Klebsiella pneumoniae isolated from children with bloodstream infection[J]. Chinese Journal of Nosoco-miology, 2021, 31(21): 3337-3341. |
| [20] |
Wu XB, Shi QY, Shen SM, et al. Clinical and bacterial characteristics of Klebsiella pneumoniae affecting 30-day mortality in patients with bloodstream infection[J]. Front Cell Infect Microbiol, 2021, 11: 688989. |



