2. 蚌埠医学院第一附属医院 检验科, 安徽 蚌埠 233004
2. Department of Laboratory Medicine, The First Affiliated Hospital of Bengbu Medical College, Bengbu 233004, China
近年来,耐碳青霉烯类肺炎克雷伯菌(carbape-nems-resistant Klebsiella pneumoniae,CRKP)在临床的检出率呈增长趋势,尤其在重症医学科,CRKP多为泛耐药菌株,治疗药物选择困难,且易引起医院感染的暴发流行,给临床诊疗及医院感染防控带来巨大的风险与挑战[1-2]。快速、准确检测CRKP对临床重症感染患者的快速诊断,及时治疗,以及减少医院感染风险,减少患者住院费用,降低感染患者病死率等具有重要意义。CRKP菌株的检测常见方法有耐药表型检测、免疫学方法检测碳青霉烯酶以及分子生物学方法检测碳青霉烯酶基因等[3],但上述方法存在检测时间长、试验条件要求高、检测费用昂贵等不足。基质辅助激光解析/电离飞行时间质谱(matrix-assisted laser desorption/ionization time-of-flight mass spectrometry,MALDI-TOF MS)通过鉴定微生物的蛋白图谱,为临床菌株的快速鉴别提供强有力的技术支持[4]。有研究[5-6]表明,MALDI-TOF MS可用于快速鉴别耐药菌,如耐甲氧西林金黄色葡萄球菌(methicillin-resistant Staphylococcus aureus,MRSA)和耐万古霉素肠球菌(vancomycin-resistant Enterococcus,VRE)。CRKP最主要的耐药机制是产生由blaKPC、blaIMP、blaNDM-1、blaVIM和blaOXA-48基因等介导的碳青霉烯酶,其中我国主要的流行株为blaKPC-2型[7]。MALDI-TOF MS快速检测CRKP的相关研究较少,大多应用BioTyper软件进行图谱分析[8]。本研究以PCR方法为“金标准”[9],应用MALDI-TOF MS和SARAMIS软件建立耐药型和敏感型2种超级图库(Super-Spectra),用于快速鉴定携带blaKPC-2基因型肺炎克雷伯菌。
1 材料与方法 1.1 材料 1.1.1 菌株来源收集2018年9月—2020年11月蚌埠医学院第一附属医院临床住院患者分离自痰、血、尿及胸腔积液等标本的肺炎克雷伯菌,排除同一患者相同部位检出的重复菌株。质控菌株:大肠埃希菌ATCC 8739、大肠埃希菌ATCC 25922、肺炎克雷伯菌ATCC BAA-1705(blaKPC-2阳性)、ATCC BAA-1706(blaKPC-2阴性)。菌株均保存于该院微生物实验室。
1.1.2 试剂及仪器MH平板、哥伦比亚血琼脂平板购自合肥天达试剂公司;MALDI-TOF MS质谱仪、48孔靶板及CHCA基质液购自法国生物梅里埃公司;基因组DNA提取试剂盒购自北京Tiangen有限公司;美罗培南(10 μg)和亚胺培南(10 μg)药敏卡片购自英国Oxoid公司;PCR试剂盒购自日本Takara公司。
1.2 方法 1.2.1 菌株培养与种属鉴定采用三区划线,接种收集的菌株,在(36±1)℃培养箱中培养18~24 h。挑取单一菌落,应用甲酸萃取法[8]进行处理,MALDI-TOF MS鉴定菌株,每个菌株两个靶点,图谱由自带的RUO系统比对获取。
1.2.2 耐药表型检测采用K-B法检测菌株耐药性,结果参照2020版美国临床和实验室标准协会(CLSI) M100 30th药敏标准执行。
1.2.3 耐药基因检测从耐药表型检测获得的CRKP菌株中,挑取单一菌落,接种于3 mL液体肉汤培养基中,进行过夜培养;将培养菌液参照说明书,应用DNA提取试剂盒,从中获得基因组DNA(OD260/OD280为1.7~1.9);根据blaKPC-2基因特异引物(见表 1),对基因组DNA进行聚合酶链反应(PCR)扩增,扩增阳性产物送至测序公司进行基因测序,结果与美国国立生物技术信息中心(NCBI) GenBank数据库对比确认。
| 表 1 耐药基因PCR扩增引物序列及产物大小 Table 1 Primer sequences and size of PCR products of resistance genes |

|
选取70株肺炎克雷伯菌,参照1.2.1方法提取检出blaKPC-2基因型肺炎克雷伯菌菌体蛋白,并进行图谱采集。结果以置信区间为衡量标准(置信区间为75%~99.9%表示结果可信,<75%表示结果不可信)。将收集的图谱保存至一个文件夹中,去除较大差异的图谱(确认Taxonomy>80%), 峰的质量(datacount)为80~230;选择质谱峰度相同的最特异的峰,确保每种质量峰至少80%的菌株拥有,最终保留35~49个峰(目标39~41),与数据库进行比对,确定权重,建立携带blaKPC基因型肺炎克雷伯菌的Super-Spectra,用于下一步的验证。碳青霉烯类敏感肺炎克雷伯菌(carbapenem-sensitive Klebsiella pneumoniae, CSKP) Super-Spectra的建立方法同上,激活后用于验证。
1.2.5 Super-Spectra验证根据1.2.1的方法,选择除建库以外的肺炎克雷伯菌,利用MALDI-TOF MS收集图谱,应用新建的Super-Spectra验证是否耐药;以K-B法和PCR结果作为鉴定标准。
2 结果 2.1 耐药表型及基因型分布共收集295株肺炎克雷伯菌,经耐药表型筛选CRKP 143株和CSKP 152株;CRKP菌株中检出携带碳青霉烯酶基因140株,经测序鉴定为携带blaKPC-2基因134株,携带blaKPC-18基因3株,携带blaNDM-1基因2株,携带blaIMP基因1株,未鉴定出blaOXA-48基因;其中2株同时检出blaKPC-2和blaNDM-1基因。质控菌株ATCC BAA-1705携带blaKPC-2基因。部分菌株PCR结果见图 1。
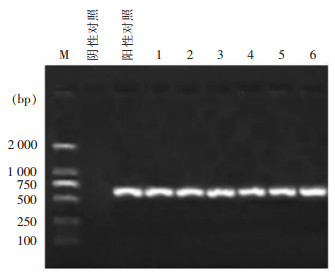 |
| M:2 000 bp DNA Marker;1~6泳道:CRKP菌株。 图 1 blaKPC-2基因扩增电泳图 Figure 1 Amplification electrophoresis map of blaKPC-2 gene |
以PCR为标准,选取其中携带blaKPC-2基因的肺炎克雷伯菌70株,并加入ATCC BAA-1705,共71株CRKP建立Super-Spectra;菌株Taxonomy均>80%,收集最特异的40个特异峰,确定权重为28,获得携带blaKPC-2基因肺炎克雷伯菌的Super-Spectra,见图 2。
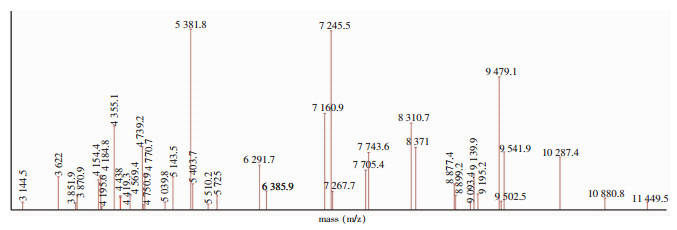 |
| 图 2 携带blaKPC-2基因肺炎克雷伯菌的Super-Spectra图谱 Figure 2 Super-Spectra map of blaKPC-2-producing K. pneumoniae |
选取CSKP 70株,加入ATCC BAA-1706,共71株,确定权重为28,建立Super-Spectra,图谱见图 3。
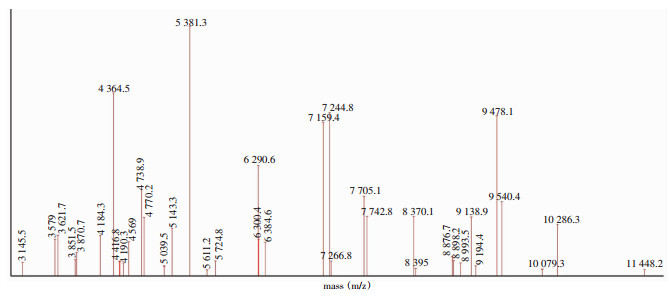 |
| 图 3 CSKP Super-Spectra图谱 Figure 3 Super-Spectra map of CSKP |
设置error值<0.05,二者的图谱重合率为80%,见图 4。比较发现,4 154.4、8 310.7、10 880.8 m/z是携带blaKPC-2基因肺炎克雷伯菌的特征峰,3 579、10 079.3 m/z是CSKP的特征峰。上述5个峰可作为区分携带blaKPC-2基因肺炎克雷伯菌和CSKP的特征峰。
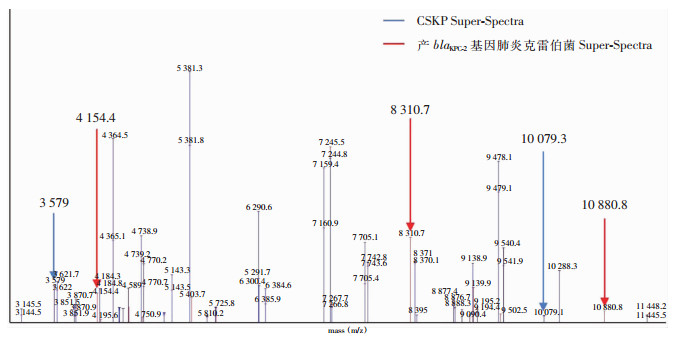 |
| 图 4 携带blaKPC-2基因肺炎克雷伯菌和CSKP的Super-Spectra对比图 Figure 4 Super-Spectra comparison of blaKPC-2-harboring K. pneumoniae and CSKP |
将除建库以外的155株肺炎克雷伯菌作为验证菌株,以K-B法和PCR作为参考标准,验证携带blaKPC-2基因肺炎克雷伯菌和CSKP的Super-Spectra图库的准确性,见表 2。携带blaKPC-2基因肺炎克雷伯菌准确率94.52%(69/73),CSKP准确率91.46%(75/82),新建的Super-Spectra鉴定肺炎克雷伯菌是否对碳青霉烯类抗生素耐药的准确率为92.90%(144/155)。
| 表 2 Super-Spectra图库验证结果 Table 2 Validation results Super-Spectra |
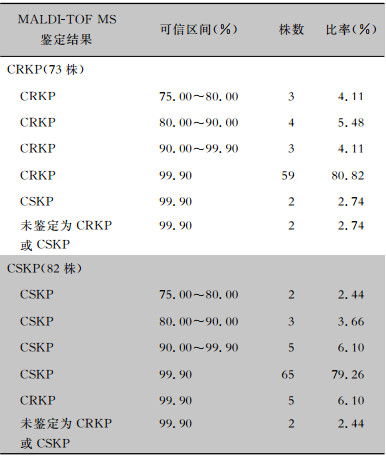
|
CRKP是医院感染的重要致病菌,感染治疗困难,患者病死率高,同时容易在医院内传播引起医院感染[10]。而检测CRKP的传统方法耗时长,分子检测手段灵敏度和特异度高,但是对试验环境以及人员要求较高,且价格昂贵。因此,快速诊断CRKP有利于临床早期正确选择抗菌药物,降低患者病死率,建立一种快速、简便且价格较低的CRKP检测方法尤为重要。
MALDI-TOF MS是近年来广泛应用于临床的快速鉴定病原菌的新方法。研究[11-12]显示MALDI-TOF MS在快速鉴别病原菌的同时可用于CRKP的检测,通过检测菌株与碳青酶烯类抗生素作用一段时间后的水解产物,或者直接检测菌株的碳青霉烯酶质谱特征峰来判断CRKP。还有研究[13]采用直接靶板微滴生长法评估菌株对碳青酶烯类抗生素的最小抑菌浓度(MIC)值从而鉴别CRKP。其中直接检测碳青霉烯酶的质谱特征峰最为便捷。MALDI-TOF MS快速鉴定微生物主要是BioTyper和SARAMIS两种主要系统,其中BioTyper检测CRKP的方法多用于科研[11, 14],需要通过两组数据的聚类分析和主成分分析,根据多菌株或多样本获得验证结果,操作繁琐,易导致容错率降低。而本次研究以鉴定单一菌株为目的,适合临床某一种菌株或亚种的初筛。本研究通过初筛出携带blaKPC-2肺炎克雷伯菌,作为建立Super-Spectra图库的试验菌株,能够鉴定大多数CRKP,可作为临床第一步快速筛选耐药菌株的有效手段。通过采用MALDI-TOF MS RUO系统和SARAMIS软件,对携带blaKPC-2基因的肺炎克雷伯菌和CSKP进行对比分析,发现区别两者的特征峰。与常规鉴定仪相比,通过质谱比对特征峰,可在5~10 min内快速初筛,具有时间短、高通量、准确性高、特异性高等优点,并且采用单一菌株,弥补了时效长、准确性低、操作繁琐等不足,对疾病诊疗及感染防控具有重要意义。本试验通过比较携带blaKPC-2基因型肺炎克雷伯菌和CSKP的超级图库,发现4 154.4、8 310.7、10 880.8 m/z是携带blaKPC-2基因型肺炎克雷伯菌的特征峰,3 579、10 079.3 m/z是CSKP的特征峰,这5个特征可以作为该院快速筛查肺炎克雷伯菌的有效工具。经过临床菌株的验证准确率为92.90%,其中携带blaKPC-2基因的肺炎克雷伯菌准确率为94.52%,CSKP准确率为91.46%。
研究显示[9, 15],不同地区碳青霉烯酶基因型分布虽有所差异,但是KPC-2型仍是多地区最常见的碳青霉烯酶。国外研究[16-17]显示,携带blaKPC-2基因型肺炎克雷伯菌的特征峰11 109 m/z,位于编码PKPQIL-p019蛋白的质粒上,但该质粒仅在研究区域内流行,在其他不携带该质粒的区域内尚未检测出该特征峰,表明菌株的遗传背景不同,其特征峰大多不尽相同。该院近3年检出的CRKP菌株也以携带KPC-2基因型为主,其他常见碳青霉烯酶基因型检出率均较低,产KPC-2型碳青霉烯酶作为CRKP菌株的主要耐药机制,且种类较为单一。依据是否存在4 154.4、8 310.7、10 880.8 m/z三个特征峰,对于本地区快速筛选携带blaKPC-2基因型肺炎克雷伯菌具有一定的临床应用价值。试验通过分析特征峰之间的差异,快速筛选耐药菌株,进行溯源性分析,判断医院内或者本地区是否存在暴发流行,具有快速、简便、经济、实用性强等优势,在区域流行病学研究中具有一定的应用价值。
本研究中有4株肺炎克雷伯菌经过质谱分析,未获得鉴定结果。耐药表型分析显示为2株耐药和2株敏感,2株耐药菌株经质谱分析,未出现4 154.4、8 310.7、10 880.8 m/z特征峰,根据测序结果发现,这2株耐药菌株同时携带blaKPC-2基因和blaNDM-1基因,两种基因之间可能存在部分干扰,导致编码蛋白出现不稳定性,干扰菌株的蛋白质表达丰度;除蛋白表达水平外,MALDI-TOF MS鉴定结果受到临床菌株来源、抗菌药物选择压力、试验室环境、操作者操作娴熟水平及待测菌株活性等因素制约;菌体可能存在不表达或表达过度,导致SARAMIS软件分析菌体的准确性不高。本次验证试验中有2株CRKP,使用质谱超级图库分析,鉴定为CSKP;5株CSKP鉴定为CRKP。可能由于基因的遗传背景不同,使菌株蛋白表达存在差异,影响验证结果。本次试验样本数量有限,后期需要扩大试验菌株,根据CRKP的耐药机制,丰富CRKP的特征峰,提高验证的准确率。
综上所述,通过严格构建携带blaKPC-2基因型肺炎克雷伯菌和CSKP的超级图谱库,建立一种快速、准确、灵敏的初筛方法用于CRKP的鉴定,有助于在区域流行病学研究中有效快速鉴定携带blaKPC-2基因型肺炎克雷伯菌,以降低其流行和暴发的风险。在后续研究中,可进一步扩展该区域的图谱库,严格遵循要求,尽可能选取更多具有代表性的图谱,纳入携带blaKPC-2基因型肺炎克雷伯菌的超级图谱库中。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Zhang R, Dong N, Huang YL, et al. Evolution of tigecyclineand colistin-resistant CRKP (carbapenem-resistant Klebsiella pneumoniae) in vivo and its persistence in the GI tract[J]. Emerg Microbes Infect, 2018, 7(1): 127. |
| [2] |
Tang X, Xiao M, Zhuo C, et al. Multi-level analysis of bacteria isolated from inpatients in respiratory departments in China[J]. J Thorac Dis, 2018, 10(5): 2666-2675. DOI:10.21037/jtd.2018.04.46 |
| [3] |
Papp JR, Rowlinson MC, O'Connor NP, et al. Accuracy and reproducibility of the Etest to detect drug-resistant Neisseria gonorrhoeae to contemporary treatment[J]. J Med Microbiol, 2018, 67(1): 68-73. DOI:10.1099/jmm.0.000651 |
| [4] |
Kim E, Kim H, Yang SM, et al. Rapid identification of Staphylococcus species isolated from food samples by matrix-assisted laser desorption/ionization time-of-flight mass spectrometry[J]. J Microbiol Biotechnol, 2019, 29(4): 548-557. DOI:10.4014/jmb.1901.01046 |
| [5] |
Shan WG, Li JP, Fang Y, et al. Rapid identification of methicillin-resistant Staphylococcus aureus (MRSA) by the Vitek MS Saramis system[J]. Curr Microbiol, 2016, 72(1): 29-32. DOI:10.1007/s00284-015-0913-8 |
| [6] |
Griffin PM, Price GR, Schooneveldt JM, et al. Use of matrix-assisted laser desorption ionization-time of flight mass spectrometry to identify vancomycin-resistant Enterococci and investigate the epidemiology of an outbreak[J]. J Clin Micro-biol, 2012, 50(9): 2918-2931. DOI:10.1128/JCM.01000-12 |
| [7] |
Li P, Liang QQ, Liu WG, et al. Convergence of carbapenem resistance and hypervirulence in a highly-transmissible ST11 clone of K. pneumoniae: an epidemiological, genomic and functional study[J]. Virulence, 2021, 12(1): 377-388. DOI:10.1080/21505594.2020.1867468 |
| [8] |
Giordano C, Barnini S. Rapid detection of colistin-resistant Klebsiella pneumoniae using MALDI-TOF MS peak-based assay[J]. J Microbiol Methods, 2018, 155: 27-33. DOI:10.1016/j.mimet.2018.11.008 |
| [9] |
Fuga B, Royer S, Campos PAD, et al. Molecular detection of class 1 integron-associated gene cassettes in KPC-2-producing Klebsiella pneumoniae clones by whole-genome sequencing[J]. Microb Drug Resist, 2019, 25(8): 1127-1131. DOI:10.1089/mdr.2018.0437 |
| [10] |
胡付品, 郭燕, 朱德妹, 等. 2019年CHINET三级医院细菌耐药监测[J]. 中国感染与化疗杂志, 2020, 20(3): 233-243. Hu FP, Guo Y, Zhu DM, et al. CHINET surveillance of bacterial resistance across tertiary hospitals in 2019[J]. Chinese Journal of Infection and Chemotherapy, 2020, 20(3): 233-243. |
| [11] |
Figueroa-Espinosa R, Costa A, Cejas D, et al. MALDI-TOF MS based procedure to detect KPC-2 directly from positive blood culture bottles and colonies[J]. J Microbiol Methods, 2019, 159: 120-127. DOI:10.1016/j.mimet.2019.02.020 |
| [12] |
余佳佳. MALDI-TOF质谱技术在肠杆菌科细菌的碳青霉烯酶快速筛查和分子流行病学中的应用研究[D]. 上海: 上海交通大学, 2018. Yu JJ. The applied research of MALDI-TOF mass spectrometry in the matter of carbapenemase rapid screening and epidemiology of Enterobacteriaceae[D]. Shanghai: Shanghai Jiao Tong University, 2018. |
| [13] |
沈佳瑾, 黄声雷, 周春妹, 等. MALDI-TOF MS直接靶板微滴生长法对耐碳青霉烯类肠杆菌的快速鉴别诊断价值[J]. 中国临床医学, 2020, 27(4): 549-553. Shen JJ, Huang SL, Zhou CM, et al. Value of rapid identification of carbapenem-resistant Enterobacteriaceae using MALDI-TOF MS-based direct-on-target microdroplet growth assay[J]. Chinese Journal of Clinical Medicine, 2020, 27(4): 549-553. |
| [14] |
Gaibani P, Galea A, Fagioni M, et al. Evaluation of matrix-assisted laser desorption ionization-time of flight mass spectrometry for identification of KPC-producing Klebsiella pneumoniae[J]. J Clin Microbiol, 2016, 54(10): 2609-2613. DOI:10.1128/JCM.01242-16 |
| [15] |
Miriagou V, Tzouvelekis LS, Rossiter S, et al. Imipenem resistance in a Salmonella clinical strain due to plasmid-mediated class A carbapenemase KPC-2[J]. Antimicrob Agents Chemother, 2003, 47(4): 1297-1300. DOI:10.1128/AAC.47.4.1297-1300.2003 |
| [16] |
Rocco VG, Intra J, Sarto C, et al. Rapid identification of carbapenemase-producing Klebsiella pneumoniae strains by matrix-assisted laser desorption/ionization-time of flight using Vitek® mass spectrometry system[J]. Eurasian J Med, 2019, 51(3): 209-213. DOI:10.5152/eurasianjmed.2019.18405 |
| [17] |
Youn JH, Drake SK, Weingarten RA, et al. Clinical perfor-mance of a matrix-assisted laser desorption ionization-time of flight mass spectrometry method for detection of certain blaKPC-containing plasmids[J]. J Clin Microbiol, 2016, 54(1): 35-42. DOI:10.1128/JCM.01643-15 |



