2. 广州中医药大学附属广州中医医院院感科, 广东 广州 510000
2. Department of Healthcare-associated Infection Management, The Affiliated TCM Hospital of Guangzhou University of Chinese Medicine, Guangzhou 510000, China
亚胺培南、美罗培南等碳青霉烯类药物对超广谱β-内酰胺酶(ESBLs)及头孢菌素酶(AmpC)具有高度稳定性,且由于抗菌药物后效应使其具有抗菌活性强、抗菌谱广的特性,被认为是治疗肠道杆菌所致重度感染的最后一道防线[1]。随着碳青霉烯类药物的广泛使用、不合理应用,以及质粒介导的碳青霉烯酶流行,耐药基因通过质粒在细菌间传播及变迁[2-3],导致耐碳青霉烯类肺炎克雷伯菌(carbape-nem-resistant Klebsiella pneumoniae,CRKP)已呈全球散布[4]。根据全国细菌耐药监测网结果报道,肺炎克雷伯菌对亚胺培南、美罗培南的耐药率分别从2014年的4.8%、4.5%上升到2019年的10.5%、10.9%,五年间上升幅度达2倍以上[5-6],耐药率上升快速。本研究对2017—2019年CRKP相关检索文献中大量数据进行统计分析,探讨近年来CRKP在我国流行的克隆及分子流行病学特征,为临床上追踪及实时监控CRKP医院感染以及防控提供实际数据支持,现将结果报告如下。
1 资料与方法 1.1 文献检索通过计算机检索中国知网(CNKI)和万方期刊数据库,检索发表时间为2017年1月1日—2019年12月31日,检索词包括耐碳青霉烯类、肺炎克雷伯菌、耐药和基因。
1.2 文献纳入及排除标准纳入标准:(1)可获得全文的公开发表期刊论文、学位论文;(2)文献所研究的肺炎克雷伯菌株在中国境内采集,且有具体收集地点;(3)文献数据可提供菌株采集地,用聚合酶链反应(PCR)方法检测耐药基因,通过多位点序列分型(MLST)分析获得ST型别。排除标准:(1)重复报道的文献;(2)综述、病例报告、会议或不能获取全文等文献;(3)文献仅提供菌株药敏分析而不能提供PCR检测的耐药基因或MLST的ST型别。
1.3 数据提取及分析根据巴斯德研究所网站(https://bigsdb.pasteur.fr/)提供的肺炎克雷伯菌MLST数据库(https://bigsdb.pasteur.fr/cgi-bin/bigsdb/bigsdb.pl?db=pubmlst_klebsiella_seqdef),将所收集文献菌株的ST型别进行比对,获得各型别的七个管家基因(gapA、infB、mdh、pgi、phoE、rpoB和tonB),将所有菌株位点信息录入BioNumerics(Version7.6.3)软件创建最小生成树。应用Microsoft Excel 2016软件统计分析并构建相对应的统计图。
2 结果 2.1 纳入文献初步检索文献202篇,根据纳入及排除标准,下载阅读全文并手工归纳,最终纳入40篇文献[7-46]。共2 094株CRKP,来自全国15个省市,其中2 094株(100%)通过PCR检出耐药基因,859株(41.02%)通过MLST检出ST型别。根据中国七大自然地理分区,本研究暂未纳入来自西北及港澳台地区的CRKP研究数据。
2.2 耐药基因及亚型检出情况提取2 094株CRKP数据分析发现,1 631株(77.89%)CRKP携带产KPC酶耐药基因,检出亚型主要为KPC-2型(1 014株);916株(43.74%)CRKP携带超广谱β-内酰胺酶(ESBLs)耐药基因,其中携带CTX-M型280株,TEM型270株,SHV型366株。215株(10.27%)CRKP携带金属β-内酰胺酶及亚胺培南(IMP)酶耐药基因,其中携带产NDM型130株,检出亚型主要为NDM-1(45株);携带产IPM型78株,检出亚型主要为IMP-4(22株);携带产VIM型7株。80株(3.82%)CRKP携带AmpC酶耐药基因,其中携带FOX 28株,DHA 45株,CMY7株;4株(0.19%)CRKP携带OXA酶耐药基因,其中2株为OXA-48亚型;97株(4.63%)CRKP携带喹诺酮类耐药基因。见表 1。
| 表 1 094株CRKP耐药基因及亚型的检出情况 Table 1 Detection result of drug resistance genes and subtypes of 2 094 CRKP strains |
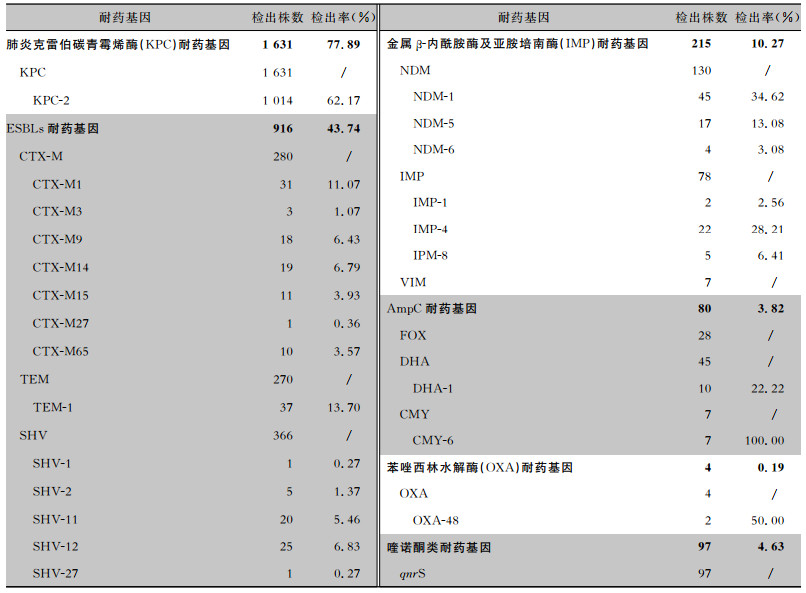
|
2 094株CRKP共检出12种耐药基因,其中KPC在各地区占比均最高,东北、华北、华东、华南、华中、西南地区分别为72.73%、92.11%、84.27%、69.39%、62.86%、91.53%。华中地区CRKP携带CTX-M(50.00%)、TEM(35.19%)、SHV(51.46%)、qnrS(23.54%)耐药基因高于其他地区。见表 2。
| 表 2 2 094株CRKP耐药基因的地区分布情况[株(%)] Table 2 Regional distribution of drug resistance genes of 2 094 CRKP strains (No. of isolates[%]) |
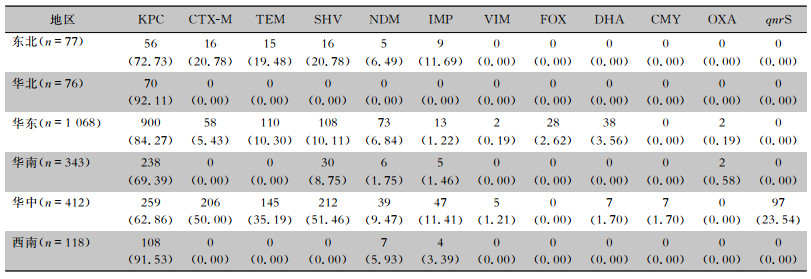
|
859株CRKP菌株进行MLST,共检出82个ST型,其中优势型别为ST11(76.72%),其次为ST15(2.44%)、ST76(1.86%)、ST23(1.28%)。见图 1。
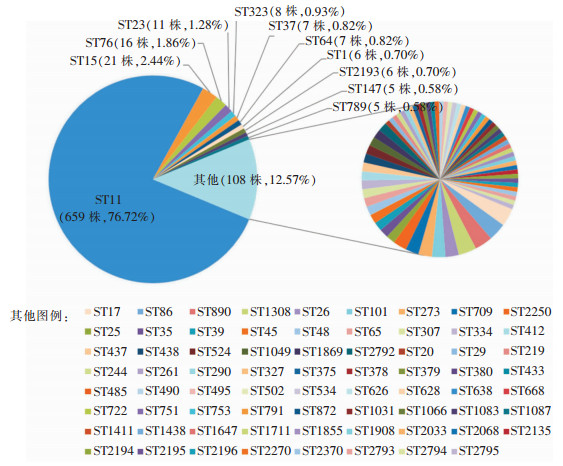 |
| 图 1 859株CRKP的ST型别检出比例 Figure 1 Detection proportion of ST of 859 CRKP strains |
859株CRKP菌株经MLST分型,共分为82种ST型,优势型别为ST11(3-3-1-1-1-1-4);65种ST型只分布在1个地区,11种ST型分布于2个地区,3种ST型分布于3个地区,1种ST型分布于4个地区,1种ST型分布于6个地区。见图 2。
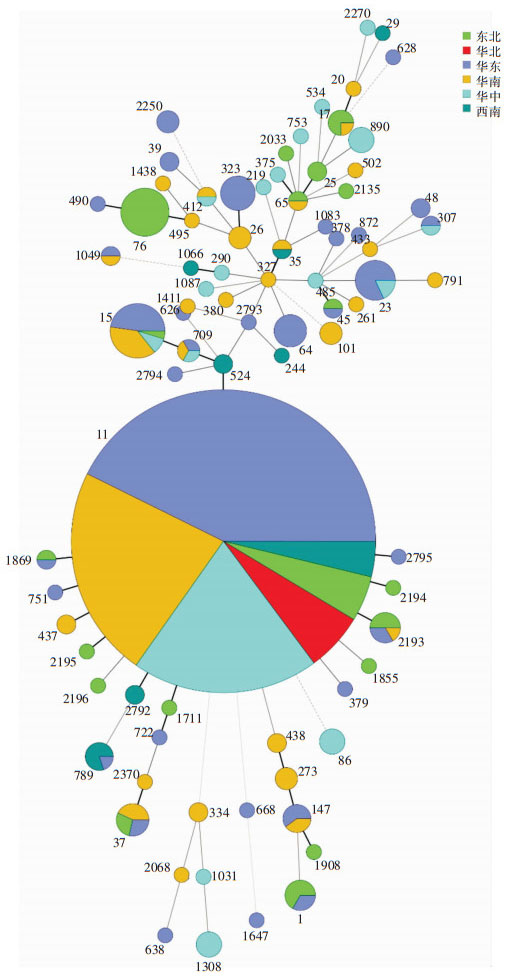 |
| 注:图中每一个圈代表一个ST型, 圈的大小代表该ST型所包含的菌株数,位点差异个数不同影响连接线由粗至细、由实至虚,最粗实线位点差异个数为1,以此类推。 图 2 859株分离自全国的CRKP MLST最小生成树 Figure 2 Minimum spanning tree for MLST of 859 CRKP strains isolated from all over the country |
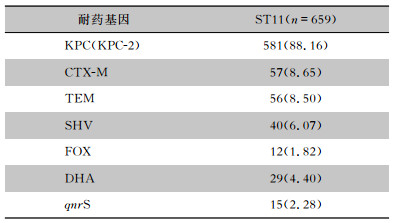
共581株CRKP携带产KPC-2酶耐药基因且MLST为ST11, 占总ST11型别(共659株)的88.16%。见表 3。
| 表 3 耐药基因与ST型别的关系[株(%)] Table 3 Relationship between drug resistance genes and ST(No. of isolates[%]) |
|
本研究纳入CRKP菌株使用PCR方法或MLST的文献,分析碳青霉烯类耐药基因以及菌株变异情况,探讨CRKP菌株的分子分型和种群结构特征。CRKP耐药机制复杂多样,研究[5, 47-48]表明,CRKP主要耐药机制为主动外排机制活跃,膜孔蛋白突变或缺失致外膜通透性降低,青霉素结合蛋白作用位点对碳青霉烯类亲和力改变,以及碳青霉烯酶的产生等。CRKP经碳青霉烯类抗生素临床治疗无效后,其耐药基因更易通过整合子、转座子等元件在细菌间进行传播,从而引起耐药菌株暴发流行[49]。CRKP菌株进行基因序列测定及同源性研究,对耐药菌株的克隆散播及医院感染的监测、防控均有非常重要的意义。
本研究创新之处在于统计分析3年CRKP相关文献数据,大数据分析发现我国CRKP主要流行的耐药基因型为KPC-2(62.17%),且在全国各地区占比均为最多,主要优势序列型别为ST11型(76.72%),在全国6个地区均有分布,与以往研究[50]结果相符,且有相关研究[51-53]报道此克隆菌株曾引起过医院感染的暴发。产KPC-2的ST11型CRKP菌株[54-56]在我国流行,且近年来逐渐被重视。既往研究[57-58]对单株CRKP菌株进行全基因组测序,阐明产KPC-2的ST11型CRKP的基因序列;另一项研究[59]对一株ST11型CRKP菌株进行Illumina结合MinION的全基因组测序,结果发现由于IS26作用在一个IncR/IncF质粒上共存3个blaKPC-2基因,IS26的多个拷贝可介导耐药基因的增殖,产生复杂的耐药基因遗传背景。已有研究[60]表明,IS26参与了blaKPC的传播和扩增;且耐碳青霉烯的高毒性肺炎克雷伯菌(CR-hvKP)更易产生KPC-2的ST11型克隆菌株,高毒力传播迅速,耐药基因很可能演变成高毒质粒[61]。
虽然对产KPC-2的ST11型CRKP耐药基因的研究不断深入,但耐药基因的散播流行迅速,据本研究显示携带此基因的耐药菌株已遍布全国6个地区。现阶段对CRKP的综合防控应建立多重耐药实时监测体系,临床严格执行抗菌药物应用指征,做到规范合理使用抗菌药物,提高医务人员医院感染防控意识,严格执行手卫生,加强无菌操作培训等措施,避免CRKP在医院内出现暴发流行。
| [1] |
Rodríguez-Baño J, Gutiérrez-Gutiérrez B, Machuca I, et al. Treatment of infections caused by extended-spectrum-beta-lactamase-, AmpC-, and carbapenemase-producing Enterobacteriaceae[J]. Clin Microbiol Rev, 2018, 31(2): e00079-17. |
| [2] |
Dolejska M, Papagiannitsis CC. Plasmid-mediated resistance is going wild[J]. Plasmid, 2018, 99: 99-111. DOI:10.1016/j.plasmid.2018.09.010 |
| [3] |
Lv LC, Wan M, Wang CZ, et al. Emergence of a plasmid-encoded resistance-nodulation-division efflux pump conferring resistance to multiple drugs, including tigecycline, in Klebsiella pneumoniae[J]. mBio, 2020, 11(2): e02930-19. |
| [4] |
Schwartz KL, Morris SK. Travel and the spread of drug-resistant bacteria[J]. Curr Infect Dis Rep, 2018, 20(9): 29. DOI:10.1007/s11908-018-0634-9 |
| [5] |
全国细菌耐药监测网. 全国细菌耐药监测网2014-2019年细菌耐药性监测报告[J]. 中国感染控制杂志, 2021, 20(1): 15-31. |
| [6] |
全国细菌耐药监测网. 全国细菌耐药监测网2014-2019年耐碳青霉烯类肺炎克雷伯菌流行病学变迁[J]. 中国感染控制杂志, 2021, 20(2): 175-179. |
| [7] |
宋国滨, 黄颖, 徐元宏. 重症监护室耐碳青霉烯类肺炎克雷伯菌的耐药机制和同源性分析[J]. 临床检验杂志, 2019, 37(4): 283-286, 292. |
| [8] |
别立翰, 马晨芸, 蒋秀娣, 等. 耐碳青霉烯类肺炎克雷伯菌耐药基因分布情况及其同源性探讨[J]. 中国医药导刊, 2019, 21(2): 110-114. DOI:10.3969/j.issn.1009-0959.2019.02.015 |
| [9] |
龚林, 刘小丽, 许慧琼, 等. 耐碳青霉烯类肺炎克雷伯菌分子流行病学研究[J]. 中国感染控制杂志, 2019, 18(7): 643-647. |
| [10] |
孙吉, 何鸽飞, 沈晖, 等. 耐碳青霉烯类肺炎克雷伯菌的药敏结果及耐药基因[J]. 中国感染控制杂志, 2019, 18(6): 489-494. |
| [11] |
胡志军, 吴希静, 潘晓龙, 等. 耐碳青霉烯类肺炎克雷伯菌的耐药基因分析[J]. 安徽医学, 2019, 40(4): 420-422. DOI:10.3969/j.issn.1000-0399.2019.04.019 |
| [12] |
黄丹艳. 浙江省碳青霉烯类抗生素耐药肺炎克雷伯菌流行特征及耐药基因分析[D]. 杭州: 浙江大学, 2017.
|
| [13] |
钟太清, 李毓龙, 刘宝涛, 等. 医院获得性血流感染来源碳青霉烯类耐药肺炎克雷伯菌耐药分析[J]. 中国实验诊断学, 2018, 22(5): 765-768. DOI:10.3969/j.issn.1007-4287.2018.05.002 |
| [14] |
付方俊, 白永凤, 程颖, 等. 血流感染耐碳青霉烯类肺炎克雷伯菌耐药基因及毒力因子研究[J]. 中国卫生检验杂志, 2018, 28(18): 2217-2219, 2222. |
| [15] |
高倩倩, 殷杏, 祝俊英, 等. 碳青霉烯类耐药肺炎克雷伯菌的分子特征[J]. 中国感染与化疗杂志, 2018, 18(1): 53-57. |
| [16] |
林佛君, 胥志超, 林志伟, 等. 碳青霉烯类耐药肺炎克雷伯菌的毒力及多位点序列研究[J]. 深圳中西医结合杂志, 2018, 28(11): 1-3. |
| [17] |
杨雪, 刘琳, 赵丹, 等. 耐碳青霉烯类肺炎克雷伯菌耐药基因检测与分子流行病学研究[J]. 重庆医学, 2018, 47(15): 1977-1980, 1985. DOI:10.3969/j.issn.1671-8348.2018.15.001 |
| [18] |
毕茹茹, 姜飞, 康海全, 等. 耐碳青霉烯类肺炎克雷伯菌耐药基因及同源性分析[J]. 临床检验杂志, 2018, 36(4): 293-296, 313. |
| [19] |
俞凤. 耐碳青霉烯类肺炎克雷伯菌临床分离株耐药机制及毒力基因的研究[D]. 南昌: 南昌大学, 2018.
|
| [20] |
张志军, 鹿麟, 牛法霞, 等. 耐碳青霉烯类肺炎克雷伯菌的耐药机制与分子流行病学特征[J]. 中国感染控制杂志, 2018, 17(9): 759-763. |
| [21] |
苏珊珊, 宫雪, 张吉生, 等. 重症监护室流行耐碳青霉烯类肺炎克雷伯菌的耐药机制及同源性分析[J]. 中国感染与化疗杂志, 2018, 18(5): 508-514. |
| [22] |
黄亚雨, 涂海健, 郑磊. 耐碳青霉烯类肺炎克雷伯菌的耐药机制及同源性分析[J]. 山东医药, 2018, 58(40): 28-31. DOI:10.3969/j.issn.1002-266X.2018.40.007 |
| [23] |
刘婷婷, 杜鸿, 周惠琴, 等. 临床耐碳青霉烯类肺炎克雷伯菌分子流行病学特征研究[J]. 中国抗生素杂志, 2018, 43(11): 1436-1442. DOI:10.3969/j.issn.1001-8689.2018.11.019 |
| [24] |
刘鹏. 江西部分医院耐碳青霉烯类肺炎克雷伯菌感染危险因素、分子流行病学及耐药基因研究[D]. 南昌: 南昌大学, 2018.
|
| [25] |
杜芳玲, 梅艳芳, 万腊根, 等. 高黏液型肺炎克雷伯菌荚膜血清分型及碳青霉烯类耐药机制研究[J]. 中国感染与化疗杂志, 2018, 18(3): 278-285. |
| [26] |
李永伟, 许晓娜, 王志盛, 等. 郑州市耐碳青霉烯类肺炎克雷伯菌耐药趋势分析[J]. 中华医院感染学杂志, 2018, 28(21): 3207-3210, 3214. |
| [27] |
周开矿, 邹杨, 毕茹茹, 等. 耐碳青霉烯类肺炎克雷伯菌的分子流行特点及耐药机制[J]. 中华医院感染学杂志, 2018, 28(5): 795-800. |
| [28] |
凌勇. 碳青霉烯类耐药肺炎克雷伯菌的临床特征及耐药机制分析[D]. 广州: 广州医科大学, 2018.
|
| [29] |
李睿, 辛力华, 张青, 等. 碳青霉烯类耐药肺炎克雷伯菌ERIC-PCR指纹图谱分型的研究[J]. 解放军医药杂志, 2018, 30(11): 102-105. DOI:10.3969/j.issn.2095-140X.2018.11.026 |
| [30] |
黄峰, 许元元. 耐碳青霉烯类肺炎克雷伯菌耐药基因检测及同源性[J]. 中国感染控制杂志, 2018, 17(1): 21-25. |
| [31] |
李玉雪, 郭玉梅. 耐碳青霉烯类肺炎克雷伯菌的耐药表型及同源性分析[J]. 中华医院感染学杂志, 2018, 28(17): 2572-2575. |
| [32] |
史玮炀, 刘洋, 韩逸超, 等. 耐碳青霉烯类肺炎克雷伯菌的分子流行病学分析[J]. 检验医学, 2018, 33(1): 37-43. |
| [33] |
孔子艳, 沈俊, 李心愿, 等. 患儿分离的碳青霉烯类耐药肺炎克雷伯菌耐药基因分析[J]. 中华医院感染学杂志, 2018, 28(20): 3124-3127, 3131. |
| [34] |
罗湘蓉, 罗力, 胡方芳, 等. 贵阳某医院耐碳青霉烯类肺炎克雷伯菌的分子流行病学分析[J]. 中国抗生素杂志, 2018, 43(5): 624-629. DOI:10.3969/j.issn.1001-8689.2018.05.024 |
| [35] |
周萍, 邓一脉, 蒋廷旺, 等. 常熟地区耐碳青霉烯类肺炎克雷伯菌耐药机制及同源性分析[J]. 临床检验杂志, 2018, 36(9): 672-674, 679. |
| [36] |
巫之韵. 碳青霉烯类耐药肺炎克雷伯菌主要流行克隆株转换的机制研究[D]. 福州: 福建医科大学, 2017.
|
| [37] |
吴娜, 田素飞, 褚云卓, 等. 碳青霉烯类耐药肺炎克雷伯菌多位点序列分型和不同ST分型感染患者的临床特点[J]. 中国感染与化疗杂志, 2017, 17(5): 509-515. |
| [38] |
周道平, 陆伟桃. 耐碳青霉烯类药物肺炎克雷伯菌耐药基因研究[J]. 实验与检验医学, 2017, 35(2): 212-214, 218. DOI:10.3969/j.issn.1674-1129.2017.02.023 |
| [39] |
殷杏. 耐碳青霉烯类肺炎克雷伯菌与高毒力肺炎克雷伯菌分子特征和毒力比较[D]. 上海: 第二军医大学, 2017.
|
| [40] |
金亮, 王启, 王术艺. 耐碳青霉烯类肺炎克雷伯菌基因检测及多位点序列分析[J]. 实用医学杂志, 2017, 33(24): 4156-4160. DOI:10.3969/j.issn.1006-5725.2017.24.034 |
| [41] |
毕颖敏, 沈震, 董栋, 等. 碳青霉烯类耐药肺炎克雷伯菌药物敏感性及blaKPC基因检出率[J]. 中国感染与化疗杂志, 2017, 17(3): 298-302. |
| [42] |
谢思. 耐碳青霉烯类肺炎克雷伯菌的耐药性及同源性研究[D]. 广州: 南方医科大学, 2017.
|
| [43] |
孔晓明, 周翔, 芮志莲, 等. 耐碳青霉烯类肺炎克雷伯菌β-内酰胺酶基因调查[J]. 中国消毒学杂志, 2017, 34(11): 1027-1029. |
| [44] |
赵鹤进, 宋晓光. 儿童碳青霉烯类耐药肺炎克雷伯菌耐药机制及同源性相关分析[J]. 中国合理用药探索, 2017, 14(8): 1-4, 9. DOI:10.3969/j.issn.2096-3327.2017.08.001 |
| [45] |
张筠, 陈冲, 王珊珊, 等. 4种β-内酰胺酶基因在一组耐碳青霉烯类肺炎克雷伯菌中的流行分析[J]. 中华医院感染学杂志, 2017, 27(24): 5530-5533. |
| [46] |
吕爽. 肺炎克雷伯菌碳青霉烯酶基因型的研究[D]. 长春: 吉林大学, 2017.
|
| [47] |
Sun KD, Chen X, Li CS, et al. Clonal dissemination of multilocus sequence type 11Klebsiella pneumoniae carbapenemase-producing K. pneumoniae in a Chinese teaching hospital[J]. APMIS, 2015, 123(2): 123-127. DOI:10.1111/apm.12313 |
| [48] |
陈捷, 朱立军, 李万翔. 肠杆菌科细菌碳青霉烯酶基因的检测[J]. 中国微生态学杂志, 2016, 28(7): 825-827. |
| [49] |
Zhao WH, Hu ZQ. Acquired metallo-β-lactamases and their genetic association with class 1 integrons and ISCR elements in Gram-negative bacteria[J]. Future Microbiol, 2015, 10(5): 873-887. DOI:10.2217/fmb.15.18 |
| [50] |
陈东科, 周海健. 北京某医院耐碳青霉烯类抗生素肺炎克雷伯菌分子流行病学研究[J]. 中华预防医学杂志, 2017, 51(10): 896-902. DOI:10.3760/cma.j.issn.0253-9624.2017.10.006 |
| [51] |
Jiang Y, Wei Z, Wang Y, et al. Tracking a hospital outbreak of KPC-producing ST11Klebsiella pneumoniae with whole genome sequencing[J]. Clin Microbiol Infect, 2015, 21(11): 1001-1007. DOI:10.1016/j.cmi.2015.07.001 |
| [52] |
Yu J, Tan K, Rong ZH, et al. Nosocomial outbreak of KPC-2- and NDM-1-producing Klebsiella pneumoniae in a neonatal ward: a retrospective study[J]. BMC Infect Dis, 2016, 16(1): 563. DOI:10.1186/s12879-016-1870-y |
| [53] |
Li J, Zou MX, Wang HC, et al. An outbreak of infections caused by a Klebsiella pneumoniae ST11 clone coproducing Klebsiella pneumoniae carbapenemase-2 and RmtB in a Chinese teaching hospital[J]. Chin Med J (Engl), 2016, 129(17): 2033-2039. DOI:10.4103/0366-6999.189049 |
| [54] |
Gu B, Bi RR, Cao XL, et al. Clonal dissemination of KPC-2-producing Klebsiella pneumoniae ST11 and ST48 clone among multiple departments in a tertiary teaching hospital in Jiangsu Province, China[J]. Ann Transl Med, 2019, 7(23): 716. DOI:10.21037/atm.2019.12.01 |
| [55] |
Yu XL, Zhang W, Zhao ZP, et al. Molecular characterization of carbapenem-resistant Klebsiella pneumoniae isolates with focus on antimicrobial resistance[J]. BMC Genomics, 2019, 20(1): 822. DOI:10.1186/s12864-019-6225-9 |
| [56] |
Chi XH, Hu GC, Xu H, et al. Genomic analysis of a KPC-2-producing Klebsiella pneumoniae ST11 outbreak from a tea-ching hospital in Shandong province, China[J]. Infect Drug Resist, 2019, 12: 2961-2969. DOI:10.2147/IDR.S221788 |
| [57] |
Xu J, Zhao Z, Ge YM, et al. Unravelling the genome sequence of NDM-1 and KPC-2 co-producing Klebsiella pneumoniae ST11 isolated from a bloodstream infection[J]. J Glob Antimicrob Resist, 2020, 20: 339-341. DOI:10.1016/j.jgar.2020.01.021 |
| [58] |
Wu XL, Han HM, Chen CL, et al. Genomic characterisation of a colistin-resistant Klebsiella pneumoniae ST11 strain co-producing KPC-2, FloR, CTX-M-55, SHV-12, FosA and RmtB causing a lethal infection[J]. J Glob Antimicrob Resist, 2019, 19: 78-80. DOI:10.1016/j.jgar.2019.08.023 |
| [59] |
Feng Y, Liu L, McNally A, et al. Coexistence of three blaKPC-2 genes on an IncF/IncR plasmid in ST11Klebsiella pneumoniae[J]. J Glob Antimicrob Resist, 2019, 17: 90-93. DOI:10.1016/j.jgar.2018.11.017 |
| [60] |
He SS, Hickman AB, Varani AM, et al. Insertion sequence IS26 reorganizes plasmids in clinically isolated multidrug-resistant bacteria by replicative transposition[J]. mBio, 2015, 6(3): e00762. |
| [61] |
Zhang YW, Jin LY, Ouyang PW, et al. Evolution of hypervirulence in carbapenem-resistant Klebsiella pneumoniae in China: a multicentre, molecular epidemiological analysis[J]. J Antimicrob Chemother, 2020, 75(2): 327-336. DOI:10.1093/jac/dkz446 |



