2. 空军军医大学西京医院护理部, 陕西 西安 710032
2. Department of Nursing, Xijing Hospital, Air Force Medical University, Xi'an 710032, China
肺炎克雷伯菌(Klebsiella pneumoniae)属于人体正常菌群,也是条件致病菌,其引起的社区获得性和医院获得性感染逐年增加,特别是高毒力肺炎克雷伯菌和多重耐药肺炎克雷伯菌的传播,给临床治疗带来极大的困难[1]。临床重症感染患者的不断增加,碳青霉烯类抗生素的广泛使用,使得耐碳青霉烯类肺炎克雷伯菌(carbapenem-resistant Klebsiella pneumoniae,CRKP)菌株检出呈逐年增多的趋势。CRKP具有在医院广泛传播的潜在风险,其引发的临床感染病死率较高,治疗药物有限,给患者、医院和社会带来极大的影响和危害[2]。多位点序列分型(multilocus sequence typing,MLST)和脉冲场凝胶电泳(pulsed-field gel electrophoresis,PFGE)是两种最常用于细菌分型的分子生物学方法,可用于分析医院分离CRKP的基因型和同源性,从切断感染途径方面入手指导、控制多重耐药菌的传播。本研究收集某医院重症监护病房(ICU)4个月内连续检出的10株CRKP,进行抗菌药物敏感性试验和碳青霉烯酶基因检测,并应用MLST和PFGE方法进行菌株同源性分析,发现其传播规律和流行特点,为医院感染暴发的有效预防提供防控依据。
1 资料与方法 1.1 菌株来源收集2017年9—12月某三甲医院ICU连续检出的CRKP。
1.2 主要仪器试剂VITEK 2 Compact全自动微生物鉴定及药敏分析系统、GN革兰阴性菌鉴定卡片、AST-GN13革兰阴性菌药敏卡片(法国Biomerieux),GeneXpert分子诊断系统、Xpert® Carba-R碳青霉烯酶基因检测试剂盒(美国Cepheid),抗菌药物药敏纸片(英国Oxoid),血琼脂培养基、M-H培养基(郑州安图生物),质控菌株大肠埃希菌ATCC 25922,肺炎克雷伯菌ATCC BAA1705和ATCC BAA1706(国家微生物菌种保藏中心)。聚合酶链式反应(PCR)扩增仪、凝胶电泳成像系统、CHEF MapperXa脉冲场凝胶电泳系统(美国Bio-Rad),限制性内切酶XbaI(中国TaKaRa),Gel Red核酸染料(美国Biotium)。
1.3 抗菌药物敏感性试验采用VITEK 2 Compact系统和配套的细菌鉴定卡片、药敏卡片进行鉴定以及相应的药敏试验,检测抗菌药物的最低抑菌浓度(MIC),采用K-B法补充检测部分药物敏感试验。药敏结果依据美国临床实验室标准化协会(CLSI)药敏试验2019年M100-29th标准判断结果。
1.4 碳青霉烯酶检测根据CLSI M100-29th肠杆菌科细菌碳青霉烯酶检测方法进行mCIM试验和eCIM试验,采用赛沛Xpert® Carba-R试剂盒检测blaKPC、blaNDM、blaVIM、blaOXA-48和blaIMP 5个碳青霉烯酶基因。
1.5 多位点序列分型(MLST)采用煮沸法提取细菌DNA,PCR扩增rpoB、gapA、mdh、pgi、phoE、infB和tonB 7个管家基因,引物参考Pasteur数据库。PCR条件:94℃ 2 min,94℃ 30 s、50℃ 1 min、72℃ 30 s共35个循环,72℃ 5 min。PCR产物经1.5%琼脂糖凝胶电泳鉴定为单一条带后,送上海生工生物公司测序。将管家基因的序列在Pasteur数据库(https://bigsdb.pasteur.fr/cgi-bin/bigsdb/bigsdb.pl?db=pubmlst_klebsiella_seqdef_public&page=sequenceQuery)进行比对,获得管家基因型别,将7个管家基因型别组合获得菌株ST型别。
1.6 脉冲场凝胶电泳(PFGE)分析参考美国疾病控制与预防中心(CDC)PulseNet提供的PFGE相关标准化操作程序,调整相关电泳条件,试验方法如下:用比浊仪调整细菌悬液浓度,达4.2~4.5麦氏浊度单位;加入1% Seakem Gold:1% SDS胶,混匀制备胶块;用含蛋白酶K的细胞裂解液CLB在54℃水浴摇床中强力震荡(170~180 r/min)消化2 h;用无菌纯水洗涤胶块2次,TE缓冲液洗涤胶块3次;加入20U XbaⅠ限制性内切酶按比例配制酶切反应体系,37℃水浴孵育6~8 h后,电泳仪中进行脉冲场电泳;电泳参数为电场强度6 V/cm,夹角120度,初始脉冲为6 s,最终脉冲为36 s,电泳时间19 h;电泳结束后,使用Gel Red核酸染料染色,在美国Bio-Rad凝胶成像系统中成像保存图片。
1.7 数据处理应用BioNumerics软件进行数据分析,选择Diec相关系数和UPGMA方法。
2 结果 2.1 一般资料共收集ICU CRKP菌株10株,分别标为1、2、3…10号菌株,其中1~8、10号菌株分离自5例(分别标为A、B、C、D、E)患者的临床感染标本,9号菌株分离自病房气压治疗仪面板。5例患者均为男性,年龄28~55岁,平均(46.2±11.3)岁;患者A、C、D痰标本,患者B血、引流液标本,以及患者E血、分泌物标本中各分离1株CRKP菌株,患者E痰标本中先后分离2株CRKP。同期ICU医生、护士的手,治疗车、监护仪、微量泵、血气分析仪、床栏、床头柜、门把手,以及护士长使用的电脑键盘等32个部位环境监测标本中均未分离出CRKP菌株。
2.2 药敏结果10株CRKP菌株对青霉素类、头孢菌素类、氨基糖苷类、氟喹诺酮类和碳青霉烯类抗菌药物均呈高水平耐药;6~8号及10号CRKP菌株对四环素类抗生素敏感,其余1~5号及9号CRKP菌株对包括替加环素在内的四环素类呈高水平耐药。1~5号及9号CRKP菌株具有相同的耐药表型,而6~8号及10号CRKP菌株具有相同的耐药表型。见表 1。
| 表 1 10株CRKP菌株对抗菌药物的敏感结果 Table 1 Antimicrobial susceptibility testing results of 10 strains of CRKP |
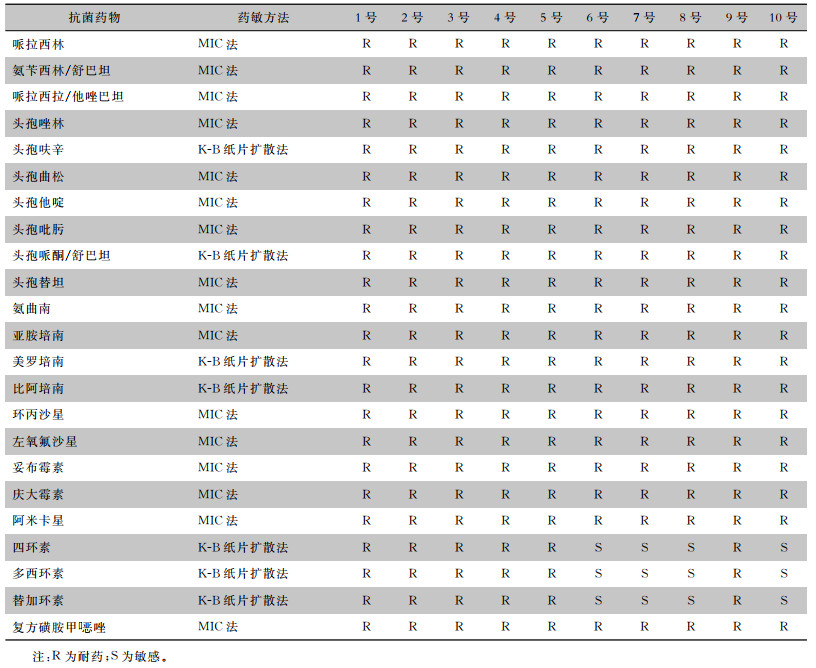
|
10株CRKP菌株mCIM试验均阳性,eCIM试验均阴性。经赛沛Xpert® Carba-R试剂盒检测,均检出blaKPC碳青霉烯酶基因。
2.4 MLST结果CRKP 7个管家基因的PCR扩增产物经1.5%琼脂糖凝胶电泳验证结果如图 1所示。PCR测序结果经DNAMAN比对、拼接,得到的序列上传到Pasteur中KPNMLST数据库进行比对分析,10株CRKP均为ST 11型。
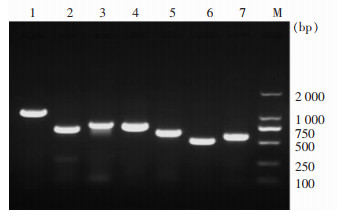 |
| M:分子量标准; 条带1~7:依次为rpoB、gapA、mdh、pgi、phoE、infB、tonB基因。 图 1 CRKP管家基因PCR扩增结果验证 Figure 1 PCR amplification results of CRKP house-keeping genes |
10株CRKP菌株经XbaI酶剪切,PFGE后得到5种带型。其中2~5号及9号CRKP菌株电泳带型完全一致,结合MLST均为ST 11型,考虑为同一来源,为流行菌株。1号CRKP菌株与流行菌株仅有1条条带的差异,两者亲缘关系接近。患者E标本中分离的6~8号和10号CRKP菌株有三种带型,与流行菌株条带相差3条以上,来自痰的6号和与来自血的10号菌株带型相同,但与同来自于痰的7号菌株带型不同。见图 2。
 |
| 图 2 10株CRKP PFGE及聚类图 Figure 2 PFGE results and cluster graph of 10 strains of CRKP |
2018年CHINET细菌耐药监测网报道,在呼吸道分离的病原菌中,肺炎克雷伯菌已经超过鲍曼不动杆菌,跃居第一位,其在血等无菌体液的分离率也逐渐升高,仅次于大肠埃希菌,位于第二[3]。CRKP的分离率也居高不下,一项来自中国的多中心研究报道,在所有耐碳青霉烯类肠杆菌科细菌(CRE)分离株中,CRKP占73.9%,耐碳青霉烯类大肠埃希菌(16.6%)和阴沟肠杆菌(7.1%)次之;在所有住院患者中,CRE感染导致的病死率高达33.5%,而CRKP在血流感染和下呼吸道感染致死率则更高[4-5]。CRKP感染主要与肺炎克雷伯菌定植或感染病史、肠外营养,使用第三代头孢菌素或碳青霉烯类抗生素有关[6]。
CRKP致死率高的原因之一是对抗菌药物广泛耐药,文献[4, 7]报道CRKP对头孢菌素类、喹诺酮类耐药率高达85%以上,对氨基糖苷类的耐药率较低,但也高达50%以上,而对革兰阴性菌感染治疗最后防线替加环素的敏感率仅有40.2%。本研究10株CRKP菌株中,6株对测试的所有抗菌药物均耐药。替加环素K-B纸片扩散法的抑菌圈仅有7 mm,表现为高水平耐药。CRKP对碳青霉烯类耐药的重要机制为产KPC酶,与国内研究[8]一致,其对替加环素耐药机制可能与外排泵表达上调等有关[9-10]。
国内流行的CRKP主要为产KPC酶的ST 11型[11-12],与本研究结果一致。但是不同医院CRKP的流行基因型不尽相同[13-14],研究CRKP的基因型别及同源性有助于监测医院感染及其发生路径,能为切断感染路径,控制医院感染提供依据。MLST和PFGE均可以用作细菌分型,且各有优劣。前者分辨率高,重复性好,数据可共享,可进行不同实验室间比对,但是测序成本高,分析缺少基因组详细信息或含有大量基因组岛插入序列,不能单独作为暴发溯源的工具;后者结果稳定,重复性好,可发现大片段的插入、缺失、重组等,酶切位点的点突变以及质粒的获得、丢失,但操作时间长,受人为影响因素大,分析的是条带而不是序列[15],通常推荐将二者结合起来进行同源性分析。
本研究中分离10株CRKP菌株的MLST均为ST 11型,未表现出菌株间差异;而通过PFGE分型得到5种带型,其中11月22—28日分离的4株(2~5号)菌株带型与12月6日于气压治疗仪面板分离的CRKP(9号)完全一致。经调查该仪器为科室共用设备,考虑本事件为气压治疗仪为媒介的接触传播导致的医院感染,该结论也表明在研究医院感染阐明菌株同源性时PFGE是MLST的有力补充。2号和3号CRKP菌株分别分离自同一重症胰腺炎患者的腹腔引流液和血标本,其PFGE带型完全一致,推测CRKP可能通过腹腔感染入血;6~8号和10号CRKP菌株分别分离自另一患者的痰、痰、引流液和血标本,只有6号与10号菌株带型完全相同,推测CRKP是通过呼吸道感染入血,同样分离自痰的6号菌株与7号菌株同源性在85%以上,可能为变异株,而分离自分泌物的8号菌株带型差异大,亲缘关系较远。以上两组数据均表明PFGE在明确细菌感染路径方面的优势。值得注意的是分离自9月16日的1号菌株与本次医院感染流行株的克隆仅差异一条条带,Tenove等[16]认为由于细菌具有变异性,在PFGE图谱分析中,相似值在0.85以上的菌株可以判定为流行病学相关,由此可见本次流行菌株可能为1号菌株变异而来。
综上所述,PFGE在明确医院感染细菌传播路径,个体细菌感染路径以及遗传变异方面具有重要应用价值。杜小莉等[17]也认为PFGE在甄别暴发菌株、高度相关菌株和不相关菌株时有很好的甄别能力,并且分型结果与菌株分离病区、分离时间具有一致性。针对暴发流行菌株,PFGE能够将相同MLST型别的菌株聚成一簇。此外,本研究也从科室环境、患者自身等多个角度发现,采取有效消毒隔离措施的重要性,尤其是患者共用设备,更需严格消毒管理,通过切断感染路径即可有效控制医院感染的发生。
| [1] |
Moradigaravand D, Martin V, Peacock SJ, et al. Evolution and epidemiology of multidrug-resistant Klebsiella pneumo-niae in the United Kingdom and Ireland[J]. mBio, 2017, 8(1): pii: e10976-16. |
| [2] |
Durante-Mangoni E, Andini R, Zampino R. Management of carbapenem-resistant Enterobacteriaceae infections[J]. Clin Microbiol Infect, 2019, 25(8): 943-950. |
| [3] |
Hu F, Guo Y, Yang Y, et al. Resistance reported from China antimicrobial surveillance network (CHINET) in 2018[J]. Eur J Clin Microbiol Infect Dis, 2019, 38(12): 2275-2281. |
| [4] |
Zhang YW, Wang Q, Yin YY, et al. Epidemiology of carbape- nem-resistant Enterobacteriaceae infections: report from the China CRE network[J]. Antimicrob Agents Chemother, 2018, 62(2): pii: e01882-17. |
| [5] |
Hauck C, Cober E, Richter SS, et al. Spectrum of excess mortality due to carbapenem-resistant Klebsiella pneumoniae infections[J]. Clin Microbiol Infect, 2016, 22(6): 513-519. |
| [6] |
Li Y, Shen H, Zhu C, et al. Carbapenem-resistant Klebsiella pneumoniae infections among ICU admission patients in central China: prevalence and prediction model[J]. Biomed Res Int, 2019, 2019: 9767313. |
| [7] |
Petrosillo N, Taglietti F, Granata G. Treatment options for colistin resistant Klebsiella pneumoniae: present and future[J]. J Clin Med, 2019, 8(7): E934. |
| [8] |
Martin J, Phan H, Findlay J, et al. Covert dissemination of carbapenemase-producing Klebsiella pneumoniae (KPC) in a successfully controlled outbreak: long- and short-read whole-genome sequencing demonstrate multiple genetic modes of transmission[J]. J Antimicrob Chemother, 2017, 72(11): 3025-3034. DOI:10.1093/jac/dkx264 |
| [9] |
Juan CH, Huang YW, Lin YT, et al. Risk factors, outcomes, and mechanisms of tigecycline-nonsusceptible Klebsiella pneumoniae bacteremia[J]. Antimicrob Agents Chemother, 2016, 60(12): 7357-7363. |
| [10] |
He T, Wang R, Liu DJ, et al. Emergence of plasmid-mediated high-level tigecycline resistance genes in animals and humans[J]. Nat Microbiol, 2019, 4(9): 1450-1456. |
| [11] |
刘婷婷, 杜鸿, 周惠琴, 等. 临床耐碳青霉烯类肺炎克雷伯菌分子流行病学特征研究[J]. 中国抗生素杂志, 2018, 43(11): 1436-1442. |
| [12] |
张志军, 鹿麟, 牛法霞, 等. 耐碳青霉烯类肺炎克雷伯菌的耐药机制与分子流行病学特征[J]. 中国感染控制杂志, 2018, 17(9): 759-763. |
| [13] |
Mshana SE, Fritzenwanker M, Falgenhauer L, et al. Molecular epidemiology and characterization of an outbreak causing Klebsiella pneumoniae clone carrying chromosomally located bla(CTX-M-15) at a German University-Hospital[J]. BMC Microbiol, 2015, 15: 122. |
| [14] |
Yan JR, Pu SL, Jia XJ, et al. Multidrug resistance mechanisms of carbapenem resistant Klebsiella pneumoniae strains isolated in Chongqing, China[J]. Ann Lab Med, 2017, 37(5): 398-407. |
| [15] |
杨晨, 胡仁静, 胡锡池, 等. MLST在碳青霉烯类耐药肺炎克雷伯菌分子流行病学分析中的应用[J]. 中华医院感染学杂志, 2016, 26(23): 5514-5516. |
| [16] |
Tenover FC, Arbeit RD, Goering RV, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis: criteria for bacterial strain typing[J]. J Clin Microbiol, 1995, 33(9): 2233-2239. |
| [17] |
杜小莉, 周海健. 肺炎克雷伯菌脉冲场凝胶电泳分型能力评价[J]. 疾病监测, 2015, 30(11): 969-975. |



