乙型肝炎病毒(hepatitis B virus, HBV)感染是一个重要的公共卫生问题。全世界约三分之一的人既往或现症感染HBV,慢性HBV携带者约3.5亿~4亿[1]。HBV感染慢性化后需要长期服抗病毒药物,往往发展为肝硬化,甚至肝细胞癌。HBV表面抗原(HBsAg)及e抗原(HBeAg)血清清除及HBV-DNA复制抑制是治疗HBV感染最重要的临床观察指标,也是慢性乙型肝炎治疗的主要目标。血清HBsAg是肝炎病毒感染的生物学标记,HBeAg阳性表示患者处于高感染低应答期,而HBV-DNA拷贝量被认为是HBV复制活跃的标志,这三个指标常用来衡量HBV感染的情况。目前,临床治疗乙型肝炎的抗病毒药物主要有干扰素、核苷(酸)类似物等。由于慢性HBV感染难根治,上述药物治疗需长期使用,致病毒耐药性逐年增加,且存在多种副作用。因此临床一线抗病毒药物的选择要求药物具有低耐药率和高病毒抑制率。新型抗HBV药物成为医学界关注点之一。
二十世纪八十年代以来,抗微生物肽(antimicrobial peptides, AMPs)也称抗菌肽,因其对感染性微生物具有抑制作用而备受关注,被认为是最有希望的抗生素替代物[2-3]。随着人们对抗菌肽研究的深入,其抗病毒作用也不断被挖掘,可通过多种机制阻止病毒感染的过程,如疱疹病毒、肝炎病毒、人类免疫缺陷病毒(HIV)等都具有包膜结构,抗菌肽通过结合病毒表面包膜并使其穿孔,从而影响病毒的繁殖、组装和释放。根据目前HBV感染及治疗的难点,结合现已发现的抗菌肽抗病毒作用,笔者模拟天然抗菌肽的结构特点,设计并合成了一系列化学合成多肽,在体外测试化学合成多肽抗HBV作用,研究不仅证明了多肽抗HBV的可行性和有效性,也为未来寻找新的HBV临床治疗药物提供思路,现将结果报告如下。
1 材料与方法 1.1 实验材料HepG 2.2.15细胞系购自中南大学湘雅医学院细胞库,该细胞株为重组载体pDoLT-HBV-1转染后的HepG 2细胞,可稳定分泌病毒颗粒和病毒蛋白[4-5],广泛应用于HBV复制相关研究。本研究所用多肽由湖南师范大学生命与科学学院容明强教授友情提供。采用传统的甲基丙烯酸聚合物平台设计7种化学合成多肽。在传甲基丙烯酸聚合物基础上设计聚合物,合成侧链上带有阳离子和疏水基团的甲基丙烯酸酯,侧链基团随机分布在聚合物链中。天然存在及既往设计的抗菌肽序列中,存在丰富的赖氨酸残基,其可发挥阳离子官能团作用,通过静电引力增强抗菌肽与细菌表面的结合。因此,本研究设计的无规共聚物以伯铵基团作为阳离子来源,模拟赖氨酸残基的阳离子功能。同时为了探索净阳离子电荷和疏水性之间的平衡,疏水基团(MPHB)的摩尔百分比(基团/阳离子多聚物)从0到60%变化。基于以上原则设计了7种多肽,并以固相法合成。序列见表 1。阳性对照药物拉米夫定购自Sigma公司(Sigma PHR1365),HBsAg检测试剂盒(ARCHITECT Anti-HBs Reagent Kit)和HBeAg检测试剂盒(ARCHITECT Anti-HBe Reagent Kit)购自雅培制药有限公司。HBV核酸定量检测试剂盒购自湖南圣湘生物科技有限公司。
| 表 1 7种化学合成多肽及序列 Table 1 Seven chemically synthesized polypeptides and their sequences |

|
将7种化学合成多肽(10 mg/mL)、拉米夫定(1 mg/mL,阳性对照)、空白溶剂(阴性对照)作用于HepG 2.2.15细胞系,检测化学合成多肽对HBV的抑制作用。
1.2.1 HBV-DNA滴度测定使用HBV核酸定量检测试剂盒对HBV-DNA进行检测。该试剂盒基于磁珠法,提取细胞上清液中的HBV-DNA,使用Taq-Man荧光探针法,在病毒核酸序列中相对保守区设计两条特异性引物、一条特异荧光探针,通过实时荧光定量PCR法(RT-PCR),于药物处理后第2、4天检测HBV-DNA。考虑到多肽可能受细胞吸收、降解等因素影响,挑选对HBV-DNA复制有抑制作用的多肽,将细胞处理时间延长至6 d及9 d以检测其抗病毒作用的稳定性。
1.2.2 HBsAg及HBeAg测定为明确化学合成多肽能否对HBsAg及HBeAg产生抑制作用,使用雅培制药有限公司HBsAg和HBeAg检测试剂盒,检测HepG 2.2.15细胞培养上清液中HBsAg及HBeAg情况(药物处理后第2、4天)。该试剂盒基于化学发光微粒子免疫检测技术,通过将样品与试剂中包被有HBs/HBe抗体的顺磁微粒子孵育,冲洗后加入吖啶脂标记,再次冲洗后依次加入预激发液及激发液,定量测量化学发光强弱,计算HBsAg和HBeAg含量。全检测流程在Abbott i2000SR仪器(雅培)中自动完成。
1.2.3 结晶紫染色法检测细胞存活率为观察化学合成多肽对细胞状态的影响,在药物培养过程中采用光镜观察细胞形态变化。同时在药物处理至9 d后,采用结晶紫染色法检测其细胞毒性作用。结晶紫可被活细胞摄取并将细胞核染成深紫色,而死亡细胞几乎不与结晶紫试剂结合,用于细胞存活检测。细胞在37℃培养并药物处理后,吸去上清培养基,PBS清洗后加入结晶紫染色液(碧云天生物技术有限公司,C0121)染色,随后弃去染色液,双蒸水充分洗涤游离染色剂,晾干后加入1% SDS溶液洗脱溶解。取100 μL于Perkinelmer EnSight多功能酶标仪中检测570 nm吸光OD值。
1.2.4 不同浓度的化学合成多肽抗HBV复制的影响为明确不同浓度的化学合成多肽对HBV-DNA复制抑制的作用,选取了HBV抑制作用最强的多肽,设置10、1、0.1 mg/mL浓度梯度,分别处理HepG 2.2.15细胞3、6、9 d后,收集细胞上清液,采用RT-PCR检测病毒DNA拷贝量,化学发光微粒子免疫分析法检测HBsAg及HBeAg的变化。
1.3 数据分析应用Prism Graphpad 5.0和SPSS 22软件对所有数据进行处理,各数据组采用t-test,Dunnet-t test,单因素重复测量方差分析等方法进行统计分析,P≤0.05为差异具有统计学意义。
| $ \begin{array}{l} {\rm{HBV - DNA}}\;{\rm{抑制率}}\;{\rm{(\% )}}\;{\rm{ = }}\\ \frac{{{\rm{(空白对照组}}\;{\rm{HBV - DNA - 实验组}}\;{\rm{HBV - DNA)}}}}{{{\rm{空白对照组}}\;{\rm{HBV - DNA}}}} \end{array} $ |
采用RT-PCR法检测细胞培养上清液中HBV-DNA拷贝量的变化。药物处理2 d时,各组药物、拉米夫定对HBV-DNA复制无明显抑制效果;药物处理4 d后,KBDT-2、5对HBV-DNA的抑制效果与拉米夫定类似,其余各组无明显抑制效果。见图 1。
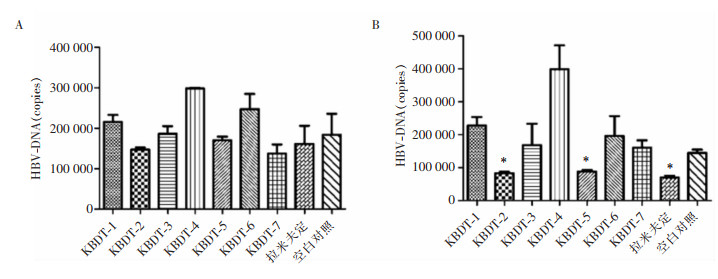 |
| A:各药物处理第2天HBV-DNA测定结果;B:各药物处理第4天HBV-DNA测定结果(*:P < 0.05) 图 1 7种化学合成多肽对HBV-DNA复制的影响 Figure 1 Effect of 7 chemically synthesized polypeptides on HBV-DNA replication |
分别使用7种化学合成多肽、拉米夫定(阳性对照)或空白溶剂(阴性对照)处理HepG 2.2.15细胞,结果显示,药物处理2 d时,KBDT-3、6、7对HBsAg有抑制效果,KBDT-1、7对HBeAg有抑制效果,其余各组无明显抑制效果;药物处理4 d时,KBDT-7对HBsAg有抑制效果,KBDT-1、7对HBeAg有抑制效果,其余各组无明显抑制效果。见图 2。
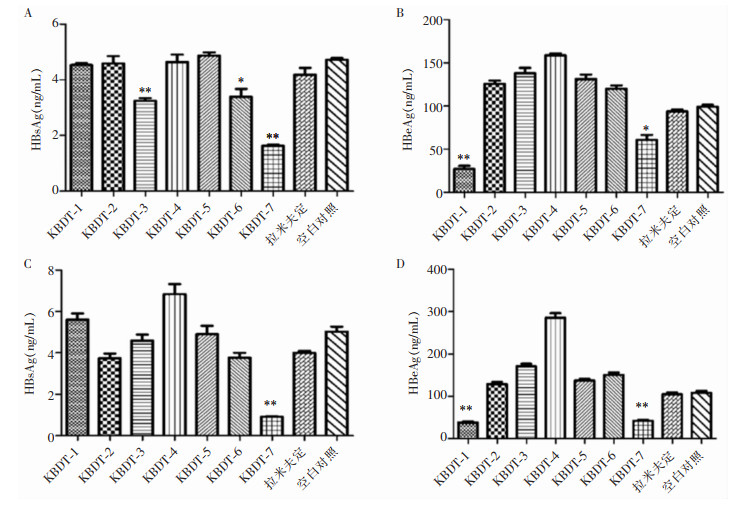 |
| A:各药物处理2 d后HBsAg测定;B:处理2 d后HBeAg测定;C:各药物处理4 d后HBsAg测定;D:处理4 d后HBeAg测定。*:P < 0.05;**:P < 0.01,n=3 图 2 化学合成多肽对HBsAg及HBeAg的影响 Figure 2 Effect of chemically synthesized polypeptides on HBsAg and HBeAg |
药物处理4 d后,镜下可见KBDT-7处理组HepG 2.2.15细胞数量明显减少,大片脱落,细胞变圆且透亮,细胞大片死亡,显示KBDT-7对细胞毒性较大。其他6种多肽、阳性对照拉米夫定及阴性对照未见明显HepG 2.2.15细胞死亡及细胞形态变化。见图 3。
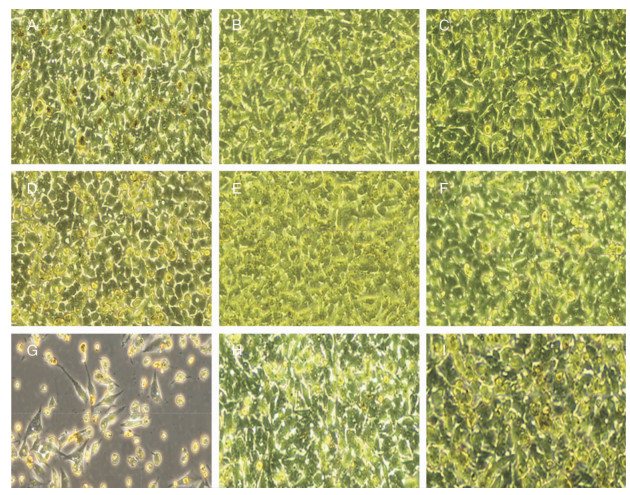 |
| 注:A~G分别为KBDT-1~7, H为拉米夫定组,I为空白溶剂处理组 图 3 经药物处理4 d后HepG 2.2.15光镜下细胞形态(100×) Figure 3 Cell morphology of HepG 2.2.15 under light microscope after 4 days of drug treatment (100 ×) |
上述实验证实,化学合成多肽KBDT-2、5可有效抑制HBV-DNA复制,将细胞处理时间延长至6 d及9 d以检测其抗病毒作用的稳定性。结果显示,处理6 d及9 d,KBDT-2、5、拉米夫定仍对HBV-DNA复制有良好抑制效果。见图 4。
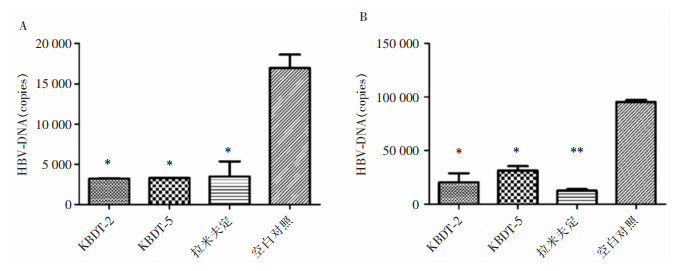 |
| A:药物处理6 d时HBV-DNA测定;B:药物处理9 d时HBV-DNA测定;*:P < 0.05;**:P < 0.01 图 4 化学合成多肽抑制HBV-DNA复制稳定性测定 Figure 4 Stability of chemically synthesized polypeptides in inhibiting HBV-DNA replication |
将多肽KBDT-2、5处理HepG 2.2.15细胞时间延长至9 d,采用结晶紫染色法检测其细胞毒性作用,结果显示处理9 d后,与空白对照比较,KBDT-2、5及拉米夫定对HepG 2.2.15无明显细胞毒性作用(P>0.05)。见图 5。
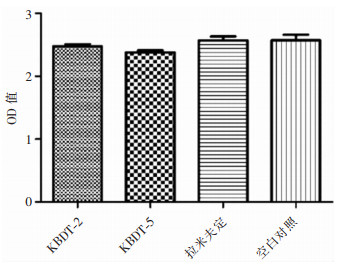 |
| 图 5 多肽KBDT-2、5处理HepG 2.2.15细胞9 d后结晶紫法检测细胞存活(OD=570 nm,n=3) Figure 5 Cell viability detected by crystal violet staining after HepG 2.2.15 cells were treated with KBDT-2 and 5 for 9 days(OD=570 nm, n=3) |
为明确不同浓度的化学合成多肽对HBV-DNA复制抑制的作用,选取HBV抑制作用最强的多肽KBDT-2,设置10、1、0.1 mg/mL浓度梯度,分别处理HepG 2.2.15细胞3、6、9 d后,收集细胞上清液,采用RT-PCR检测病毒DNA拷贝量。结果显示药物处理后,10 mg/mL KBDT-2、拉米夫定对HBV-DNA复制有抑制效果;比较10 mg/mL的KBDT-2处理3、6、9 d HBV-DNA抑制率分别为36.0%、51.5%、56.3%,抑制效果在第6天达到高峰,单因素重复测量方差分析显示,延长处理9 d后仍然维持较高抑制率,不随时间延长继续升高(F=1.240,P=0.381)。其余各组对HBV-DNA复制无明显抑制效果。见图 6。
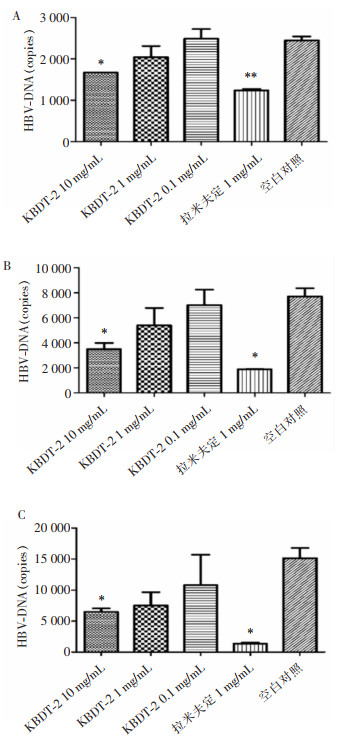 |
| A:药物处理第3天时HBV-DNA测定;B:药物处理第6天时HBV-DNA测定;C:药物处理第9天时HBV-DNA测定(Dunnet-t test,*:P < 0.05;**:P < 0.01) 图 6 不同浓度梯度的KBDT-2对HBV-DNA复制影响 Figure 6 Effect of different concentration gradients of KBDT-2 on HBV-DNA replication |
为明确不同浓度的KBDT-2对HBsAg及HBeAg的作用,同样处理后收集细胞上清液,采用化学发光微粒子免疫分析法检测HBsAg及HBeAg的变化, KBDT-2处理9 d时,10 mg/mL KBDT-2对HBsAg、HBeAg有抑制效果,其余各组无明显抑制效果。见图 7。
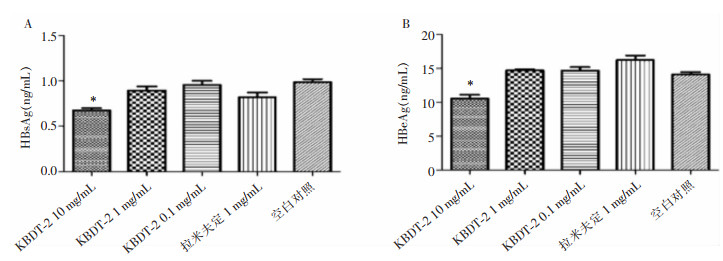 |
| A:药物处理9 d后HBsAg测定;B:药物处理9 d后HBeAg测定(*:P < 0.05,n=3) 图 7 KBDT-2对细胞培养上清中HBsAg及HBeAg的影响 Figure 7 Effect of KBDT-2 on HBsAg and HBeAg in cell culture supernatant |
采用结晶紫染色法检测不同浓度的化学合成多肽KBDT-2对细胞毒性作用,结果显示,处理9 d后,各浓度的KBDT-2及拉米夫定对HepG 2.2.15无明显毒性作用,见图 8。KBDT-2(10、1、0.1 mg/mL)、拉米夫定以及空白溶剂处理HepG 2.2.15细胞9 d后,结晶紫法检测细胞存活相比,各组差异无统计学意义(P>0.05)。
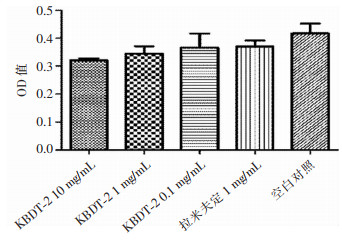 |
| 图 8 不同浓度KBDT-2处理HepG 2.2.15细胞9 d后结晶紫法检测细胞存活(OD=570 nm,n=3) Figure 8 Cell viability detected by crystal violet staining after HepG 2.2.15 cells were treated with different concentrations of KBDT-2 for 9 days(OD=570 nm, n=3) |
HBV是一种严重威胁世界各国人民健康的传染病,宿主细胞中长期的HBV活动,激活多种信号传导通路及激酶,如NF-κB、PI3K-Akt、SAPK/JNK、Jak1-STAT、MAPK[6],激活促癌基因[7],抑制抑癌基因[8], 改变细胞遗传学[9]和表观遗传学性状[10]等多种途径,导致肝细胞癌的发生[11-12],因此,有效控制慢性HBV复制是控制慢性乙肝病情进展、恶化的关键因素。
目前,临床主要抗病毒药物包括干扰素与核苷类似物。由于慢性HBV感染难根治,上述药物治疗需长期使用,存在不良反应及副作用,病毒耐药,停药后复发等不足[13]。研发新型、高效的抗HBV药物可以丰富抗HBV的“武器库”,对促进HBV感染的治疗具有积极作用。
抗菌肽广泛存在于自然界各种微生物、植物和动物(无脊椎动物和脊椎动物)中,具有抑制和杀灭细菌、真菌、包膜病毒等多种微生物的作用。抗菌肽作为潜在的“新型抗生素”,具有许多天然的优点,如相对较小的分子量(小于25 kDa~30 kDa)、两亲性结构(亲水亲脂)、带电阳离子电荷等,有利于其结合并作用于微生物;同时由于其天然存在于人体内,因此,对真核细胞杀伤作用小,副作用少,产生微生物耐药性的可能性较低。近期研究发现,通过模拟天然存在的抗菌肽, 设计并合成的化学类似物,在体外测试中展现出抗微生物的活性。如树枝状聚合物是基于丙烯酸酯单体的超分枝聚合物[14-15],在克服收率低和纯化难问题的同时,其高分子量和多价结合的特性可通过空间屏蔽或竞争性抑制,干扰病毒-宿主细胞相互作用,从而产生抗病毒活性[16]。另如基孔肯雅病毒(CHIKV,基孔肯雅热的病原体)正链RNA转录翻译的nsP2蛋白酶对病毒复制至关重要。通过电脑设计针对nsP2蛋白酶结构的多肽,从而抑制nsP2的活性,抑制CHIKV复制[17]。因此,设计并合成的破坏病毒结构或抑制关键酶类活性的多肽,可干扰病毒复制或感染过程,从而达到抗病毒的目的。抗菌肽的出现为病毒感染治疗提供了新的选择[18]。最新研究[19]表明,哺乳动物、植物、昆虫甚至化学合成来源的抗菌肽,可对HIV、疱疹病毒等产生抑制作用。与目前的抗病毒药物相比,这类多肽具有低耐药性、快速起效、限制感染扩散等优点[20],因此,本研究探索化学合成多肽对HBV复制抑制作用,可为慢性乙肝治疗提供新思路。
本研究利用传统的甲基丙烯酸聚合物平台设计7种化学合成多肽,将7种化学合成多肽作用于HepG 2.2.15细胞系,研究结果显示,化学合成多肽KBDT-2具有体外抗HBV-DNA复制作用, 呈剂量依赖。首先,KBDT-2可抑制HBsAg、HBeAg病毒基因表达,但变化时间晚于HBV-DNA降低。推测这种趋势可能是由于病毒DNA复制受抑制后,HBsAg、HBeAg转录或翻译过程滞后导致的抑制作用延迟。其次,KBDT-7虽然对HBV-DNA等检测指标具有较强抑制作用,但伴随着大量细胞的死亡。与之形成鲜明对比的是,KBDT-2在发挥抗HBV作用的同时对HepG 2.2.15无明显毒性,提示KBDT-2抗菌肽抗病毒作用并非通过对宿主细胞杀伤而起作用,可能通过某种未知机制作用于病毒复制过程,然而具体机制仍然有待进一步研究。根据已有报道,多肽可能通过影响DNA、RNA聚合酶、逆转录酶活性,或通过影响病毒复制过程中的蛋白合成以及病毒组装、成熟、释放等过程而发挥抗病毒作用效果[21];另一方面,多肽也可能依赖负电荷静电力,与宿主细胞膜中糖胺聚糖相互作用[22],对细胞外病毒产生竞争反应,从而抑制病毒与宿主细胞膜结合;最后多肽也可结合并破坏病毒包膜,造成病毒颗粒不稳定[23],从而阻断病毒的感染和复制。
本研究取得了令人鼓舞的测试结果,但仍存在不足。首先,本研究仅对化学合成多肽对病毒复制,HBV生物学标志蛋白的作用,细胞毒性等进行了观察研究,但对其中的分子机制深入挖掘不足。其次,本研究仅对化学合成多肽进行了小规模、小样本的体外细胞学研究,未进行动物实验及其他临床实验,在活体中的抗HBV作用效果及毒副作用有待进一步证实。最后,抗菌肽虽然展现了一定的抗HBV作用,但与传统药物相比,其有效性仍有待提高。值得一提是,鉴于多肽分子结构的复杂性,化学合成多肽的抗HBV作用仍然具有较大优化的空间。
综上所述,本研究在分析天然抗菌肽结构特点的基础上,优化合成了多条多肽,并测试了其抗HBV的效果,是对传统意义上抗菌肽“抗细菌”作用的拓展、挖掘。同时与传统抗HBV药物相比,抗菌肽具有低耐药率、起效快、限制病原菌扩散、中和毒素、增强抗菌作用效果等诸多优势,是一种从新的机制出发抑制HBV的可能途径。因此,本研究不仅筛选出具有抑制HBV复制作用的化学合成多肽KBDT-2,也为开发新型的抗HBV药物,改善HBV临床耐药提供实验数据支持。
致谢: 感谢湖南师范大学生命科学学院容明强教授友情设计并提供抗菌肽。
| [1] |
Ott JJ, Stevens GA, Groeger J, et al. Global epidemiology of hepatitis B virus infection:New estimates of age-specific HBsAg seroprevalence and endemicity[J]. Vaccine, 2012, 30(12): 2212-2219. DOI:10.1016/j.vaccine.2011.12.116 |
| [2] |
Zasloff M. Magainins, a class of antimicrobial peptides from Xenopus skin:isolation, characterization of two active forms, and partial cDNA sequence of a precursor[J]. Proc Natl Acad Sci U S A, 1987, 84(15): 5449-5453. DOI:10.1073/pnas.84.15.5449 |
| [3] |
Wang G, Li X, Wang Z. APD2:the updated antimicrobial peptide database and its application in peptide design[J]. Nucleic Acids Res, 2009, 37(Database issue): D933-D937. |
| [4] |
Zhu X, Xie C, Li YM, et al. TMEM2 inhibits hepatitis B virus infection in HepG 2 and HepG 2.2.15 cells by activating the JAK-STAT signaling pathway[J]. Cell Death Dis, 2016, 7(6): e2239. DOI:10.1038/cddis.2016.146 |
| [5] |
Wang J, Li N, Huang ZB, et al. HBx regulates transcription factor PAX8 stabilization to promote the progression of hepatocellular carcinoma[J]. Oncogene, 2019, 38: 6696-6710. DOI:10.1038/s41388-019-0907-2 |
| [6] |
Han B, Wu J. DcR3 protects islet beta cells from apoptosis through modulating Adcyap1 and Bank1 expression[J]. J Immunol, 2009, 183(12): 8157-8166. DOI:10.4049/jimmunol.0901165 |
| [7] |
Fu S, Wang J, Hu X, et al. Crosstalk between hepatitis B virus X and high-mobility group box 1 facilitates autophagy in hepatocytes[J]. Mol Oncol, 2018, 12(3): 322-338. |
| [8] |
Chen BC, Wu WT, Ho FM, et al. Inhibition of interleukin-1 beta-induced NF-kappa B activation by calcium/calmodulin-dependent protein kinase kinase occurs through Akt activation associated with interleukin-1 receptor-associated kinase phosphorylation and uncoupling of MyD88[J]. J Biol Chem, 2002, 277(27): 24169-24179. DOI:10.1074/jbc.M106014200 |
| [9] |
Huang YK, Fan XG, Qiu F, et al. Genomics of hepatitis B virus-related hepatocellular carcinoma and adjacent noncancerous tissues with cDNA microarray[J]. Chin Med J (Engl), 2011, 124(13): 2057-2064. |
| [10] |
Yu S, Li N, Huang Z, et al. A novel lncRNA, TCONS_00006195, represses hepatocellular carcinoma progression by inhibiting enzymatic activity of ENO1[J]. Cell Death Dis, 2018, 9(12): 1184. DOI:10.1038/s41419-018-1231-4 |
| [11] |
Chan SL, Wong VW, Qin S, et al. Infection and cancer:the case of hepatitis B[J]. J Clin Oncol, 2016, 34(1): 83-90. DOI:10.1200/JCO.2015.61.5724 |
| [12] |
Fu S, Zhou RR, Li N, et al. Hepatitis B virus X protein in liver tumor microenvironment[J]. Tumour Biol, 2016, 37(12): 15371-15381. DOI:10.1007/s13277-016-5406-2 |
| [13] |
陈若蝉, 刘振国, 范学工. DAA与HCC——丙肝治疗新时代的挑战[J]. 中国感染控制杂志, 2018, 17(5): 369-372. DOI:10.3969/j.issn.1671-9638.2018.05.001 |
| [14] |
McCarthy TD, Karellas P, Henderson SA, et al. Dendrimers as drugs:discovery and preclinical and clinical development of dendrimer-based microbicides for HIV and STI prevention[J]. Mol Pharm, 2005, 2(4): 312-318. DOI:10.1021/mp050023q |
| [15] |
Toms S, Carnachan SM, Hermans IF, et al. Poly ethoxy ethyl glycinamide (PEE-G) dendrimers:dendrimers specifically designed for pharmaceutical applications[J]. ChemMedChem, 2016, 11(15): 1583-1586. DOI:10.1002/cmdc.201600270 |
| [16] |
Pini A, Giuliani A, Falciani C, et al. Antimicrobial activity of novel dendrimeric peptides obtained by phage display selection and rational modification[J]. Antimicrob Agents Chemother, 2005, 49(7): 2665-2672. DOI:10.1128/AAC.49.7.2665-2672.2005 |
| [17] |
Das PK, Puusepp L, Varghese FS, et al. Design and validation of novel chikungunya virus protease inhibitors[J]. Antimicrob Agents Chemother, 2016, 60(12): 7382-7395. |
| [18] |
Ahmed A, Siman-Tov G, Hall G, et al. Human antimicrobial peptides as therapeutics for viral infections[J]. Viruses, 2019, 11(8), pii: E704.
|
| [19] |
黎观红, 洪智敏, 贾永杰, 等. 抗菌肽的抗菌作用及其机制[J]. 动物营养学报, 2011, 23(4): 546-555. DOI:10.3969/j.issn.1006-267x.2011.04.004 |
| [20] |
Sierra JM, Fuste M, Rabanal F, et al. An overview of antimicrobial peptides and the latest advances in their development[J]. Expert Opin Biol Ther, 2017, 17(6): 663-676. DOI:10.1080/14712598.2017.1315402 |
| [21] |
Wilson SS, Wiens ME, Smith JG. Antiviral mechanisms of human defensins[J]. J Mol Biol, 2013, 425(24): 4965-4980. DOI:10.1016/j.jmb.2013.09.038 |
| [22] |
Joo HS, Fu CI, Otto M. Bacterial strategies of resistance to antimicrobial peptides[J]. Philos Trans R Soc Lond B Biol Sci, 2016, 371(1695), pii: 20150292.
|
| [23] |
Wilmes M, Cammue BP, Sahl HG, et al. Antibiotic activities of host defense peptides:more to it than lipid bilayer perturbation[J]. Nat Prod Rep, 2011, 28(8): 1350-1358. DOI:10.1039/c1np00022e |



